تعدد أشكال النوكليوتيدات المفردة
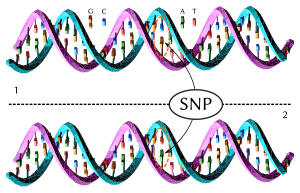
تعدد أشكال النوكليوتيد المفردة (بالإنجليزية: Single-nucleotide polymorphism أو SNP) (SNP /snɪp/ ؛ الجمع /snɪps/) هو اختلاف في سلسلة الدنا يحدث عادة في مجموعة من السكان (مثلاً %1) حيث يختلف نوكليوتيد واحد - A، T، C أو G - في المجين بين فردين من نفس النوع البيولوجي أو بين كروموسومات مزدوجة.
على سبيل المثال، مقطعي الدنا من شخصين مختلفين، AAGCCTA و AAGCTTA تحتوي على اختلاف في نوكليوتيد واحد. في هذه الحالة نقول أنه هناك أليلان. تقريبًا جميع تعدد أشكال النوكليوتيد المفرد لديها أليلين فقط. توزيع تعدد أشكال النوكليوتيد المفرد في المجين ليس متجانس; يحدث تعدد أشكال النوكليوتيد المفرد في مناطق غير الترميز أكثر من مناطق الترميز؛ أو بشكل عام، حيث يتصرف الانتقاء الطبيعي و«تصلح» أليل (مزيلة أشكال أخرى) تعدد أشكال النوكليوتيد المفرد الذي يشكّل التكيّف الوراثي الأكثر ملائمة.[1] تلعب كذلك عوامل أخرى مثل التأشيب الجيني ووتيرة الطفرات دورًا في تحديد كثافة تعدد أشكال النوكليوتيدت المفردة.[2]
في علم الوراثة، تعدد الأشكال أحادي النوكليوتيدات هو استبدال لنيوكليوتيد واحد في موضع معين في الجينوم، وهو موجود في جزء كبير بما فيه الكفاية من عدد السكان (على سبيل المثال 1٪ أو أكثر).[3]
على سبيل المثال، في موضع أساسي معين في الجينوم البشري، قد يظهر النوكليوتيد C في معظم الأفراد، ولكن في أقلية من الأفراد، يشغل هذا المنصب A. هذا يعني أن هناك SNP في هذا الموضع المحدد، ويقال أن التنوعين المحتملين للنيوكليوتيدات - C أو A - هما الأليلات لهذا الموضع المحدد.
تحدد النيوكلوتايد SNPs الاختلافات في قابليتنا للإصابة بمجموعة واسعة من الأمراض (مثل فقر الدم المنجلي والثلاسيميا بيتا والتليف الكيسي الناتج عن تعدد الأشكال).[4][5][6] تعد شدة المرض وطريقة استجابة الجسم للعلاج من مظاهر الاختلافات الجينية. على سبيل المثال، ترتبط الطفرة أحادية القاعدة في جين APOE (صميم البروتين الشحمي E) بانخفاض خطر الإصابة بمرض الزهايمر.[7]
المتغير أحادي النوكليوتيدات (SNV) هو تباين في نيوكليوتيد واحد دون أي قيود على التردد. يختلف SNV عن SNP لأنه عندما يتم اكتشاف SNV في عينة من كائن حي من نوع ما، فمن المحتمل أن يكون SNV SNP ولكن لا يمكن تحديد ذلك من كائن حي واحد فقط.[8][9] ومع ذلك، فإن SNP يعني أن النيوكليوتيدات تختلف في مجموعة الكائنات الحية. قد تنشأ SNVs في الخلايا الجسدية. يمكن أيضًا تسمية الاختلاف الجسدي أحادي النوكليوتيدات (على سبيل المثال، الناجم عن السرطان) باسم تغيير النوكليوتيدات المفردة . تظهر SNVs بشكل شائع في التشخيص الجزيئي. على سبيل المثال، عند تصميم بادئات تفاعل البوليميراز المتسلسل للكشف عن الفيروسات، قد يحتوي الحمض النووي الريبي الفيروسي أو الحمض النووي في عينة مريض واحدة على SNVs.
أنواع[عدل]
قد يقع تعدّد أشكال النوكليوتيد المفرد ضمن تسلسلات الترميز في الجينات، أو مناطق غير الترميز، أو في المناطق بين الجينات. تعدد ألأشكال داخل تسلسل الترميز لا يغيّر بالضرورة تسلسل الأحماض الأمينية في البروتين الناتج، وذلك بسبب صفة الانحلالية في الشفرة الوراثية.
| أنواع النيوكلوتايد |
|---|
وحيدة النكليوتيد تعدد الأشكال قد تدخل في الترميز تسلسل الجينات، مناطق الجينات غير الترميز، أو في المناطق بين الجينات (المناطق بين الجينات). لا تغير النيوكلوتايد ضمن تسلسل تشفير بالضرورة تسلسل الأحماض الأمينية للبروتين الذي يتم إنتاجه، بسبب انحطاط الكود الجيني.
تنقسم SNPs في منطقة الترميز إلى نوعين: SNPs مترادفة وغير مترادفة. لا تؤثر SNPs المترادفة على تسلسل البروتين، بينما تغير SNPs غير المترادفة تسلسل الأحماض الأمينية للبروتين. إن SNPs غير المترادفة من نوعين: خطأ طفرة هراء.
قد لا تزال النيوكلوتايد غير الموجودة في مناطق ترميز البروتين تؤثر على الربط الجيني أو ارتباط عامل النسخ أو تدهور الرنا المرسال أو تسلسل الحمض النووي الريبي غير المشفر. يشار إلى التعبير الجيني المتأثر بهذا النوع من SNP على أنه eSNP (تعبير SNP) وقد يكون في المنبع أو المصب من الجين.
التطبيقات[عدل]
- يمكن لدراسات الجمعية تحديد ما إذا كان المتغير الجيني مرتبطًا بمرض أو سمة.[10]
- علامة SNP عبارة عن تعدد أشكال أحادي النوكليوتيدات تمثيلي في منطقة من الجينوم مع اختلال توازن عالي الارتباط (الارتباط غير العشوائي للأليلات في موقعين أو أكثر). تعد Tag SNPs مفيدة في دراسات ارتباط SNP للجينوم الكامل، حيث يتم التنميط الجيني لمئات الآلاف من SNPs عبر الجينوم بأكمله.
- رسم خرائط النمط الفردي: يمكن تجميع مجموعات من الأليلات أو تسلسلات الحمض النووي بحيث يتمكن SNP الفردي من تحديد العديد من تعدد الأشكال المرتبطة.
- يشير اختلال التوازن (LD)، وهو مصطلح يستخدم في علم الوراثة السكانية، إلى ارتباط غير عشوائي للأليلات في موقعين أو أكثر، وليس بالضرورة على نفس الكروموسوم. يشير إلى ظاهرة تميل أن يتم توريث أليل SNP أو تسلسل الحمض النووي المتقاربين في الجينوم معًا. يمكن أن تتأثر صعوبة التعلم بمعلمين (من بين عوامل أخرى، مثل التقسيم الطبقي للسكان): 1) المسافة بين الأشكال المتعددة الأشكال [كلما كانت المسافة أكبر، كلما انخفض LD]. 2) معدل إعادة التركيب [كلما انخفض معدل إعادة التركيب، زاد LD].[11]
تكرار[عدل]
تم العثور على أكثر من 335 مليون SNPs عبر البشر من مجموعات سكانية متعددة. يختلف الجينوم النموذجي عن الجينوم البشري المرجعي في 4 إلى 5 ملايين موقع، معظمها (أكثر من 99.9٪) تتكون من تعدد الأشكال (SNPs) وindels القصيرة.[12]
داخل الجينوم[عدل]
التوزيع الجيني للنيوكليوتيدات SNPs ليس متجانسًا؛ تحدث النيوكلوتايد في المناطق غير المشفرة بشكل متكرر أكثر من المناطق المشفرة أو، بشكل عام، حيث يعمل الانتقاء الطبيعي و «يثبت» الأليل (استبعاد المتغيرات الأخرى) من SNP الذي يشكل التكيف الجيني الأكثر ملاءمة.[13] يمكن لعوامل أخرى، مثل إعادة التركيب الجيني ومعدل الطفرات، أن تحدد كثافة SNP.[14]
يمكن التنبؤ بكثافة SNP من خلال وجود سواتل مكروية: السواتل الدقيقة AT على وجه الخصوص هي مؤشرات قوية لكثافة SNP، مع وجود مساحات تكرار طويلة (AT) (n) في مناطق ذات كثافة SNP منخفضة بشكل كبير ومحتوى GC منخفض.[15]
ضمن السكان[عدل]
هناك اختلافات بين البشر، لذلك قد يكون أليل SNP الشائع في مجموعة جغرافية أو عرقية أكثر ندرة في مجموعة أخرى. ضمن مجموعة سكانية، يمكن تخصيص تواتر أليل ثانوي لـ SNPs - أدنى تردد أليل في موضع لوحظ في مجتمع معين.[16] هذا هو ببساطة أقل ترددين من الأليل لتعدد الأشكال أحادي النوكليوتيدات.
مع هذه المعرفة، طور العلماء طرقًا جديدة في تحليل الهياكل السكانية في الأنواع الأقل دراسة.[17][18][19] باستخدام تقنيات التجميع، يتم تخفيض تكلفة التحليل بشكل كبير.[بحاجة لمصدر] هذه التقنيات على تسلسل مجتمع ما في عينة مجمعة بدلاً من تسلسل كل فرد داخل المجتمع بنفسه. مع أدوات المعلوماتية الحيوية الجديدة، هناك إمكانية للتحقيق في بنية السكان وتدفق الجينات وهجرة الجينات من خلال مراقبة ترددات الأليل داخل السكان بالكامل. مع هذه البروتوكولات، هناك إمكانية للجمع بين مزايا SNPs وعلامات القمر الصناعي الصغيرة.[20][21] ومع ذلك، هناك معلومات مفقودة في العملية مثل عدم توازن الارتباط ومعلومات الزيجوزية.
أهمية[عدل]
يمكن أن تؤثر الاختلافات في تسلسل الحمض النووي للبشر على كيفية تطور البشر للأمراض والاستجابة لمسببات الأمراض والمواد الكيميائية والأدوية واللقاحات والعوامل الأخرى. تعد SNPs ضرورية أيضًا للطب الشخصي.[22] تشمل الأمثلة الأبحاث الطبية الحيوية، والطب الشرعي، وعلم الوراثة الدوائية، وسببية المرض، على النحو المبين أدناه.
الأبحاث السريرية[عدل]
تكمن الأهمية الكبرى لـ SNPs في البحث السريري في مقارنة مناطق الجينوم بين المجموعات (مثل الأفواج المتطابقة مع وبدون مرض) في دراسات الارتباط على مستوى الجينوم. تم استخدام SNPs في دراسات الارتباط على مستوى الجينوم كعلامات عالية الدقة في رسم خرائط الجينات المتعلقة بالأمراض أو السمات الطبيعية.[23] لا تزال النيوكلوتايد التي ليس لها تأثير ملحوظ على النمط الظاهري (ما يسمى الطفرات الصامتة) مفيدة كواسمات جينية في دراسات الارتباط على مستوى الجينوم، بسبب كميتها واستقرار وراثتها عبر الأجيال.[24]
التحاليل الجنائية[عدل]
تم استخدام النيوكلوتايد في البداية لمطابقة عينة من الحمض النووي الشرعي مع المشتبه به ولكن تم التخلص منها تدريجياً مع تطوير تقنيات بصمة الحمض النووي القائمة على STR.[25] قد تسمح تقنيات تسلسل الجيل التالي (NGS) الحالية باستخدام أفضل للتنميط الجيني SNP في تطبيق الطب الشرعي طالما يتم تجنب المواقع الإشكالية.[26] في المستقبل، يمكن استخدام تعدد الأشكال في الطب الشرعي لبعض القرائن النمطية مثل لون العين، ولون الشعر، والعرق، وما إلى ذلك. كيد وآخرون. أظهر أن مجموعة من 19 تعدد الأشكال يمكن أن تحدد المجموعة العرقية ذات الاحتمالية الجيدة للمطابقة (Pm = 10 −7) في 40 مجموعة سكانية تمت دراستها.[27] أحد الأمثلة على كيف يمكن أن يكون هذا مفيدًا في مجال إعادة البناء الفني لمظاهر محتملة قبل الوفاة لبقايا هيكل عظمي لأفراد مجهولين. على الرغم من أن إعادة بناء الوجه يمكن أن تكون دقيقة إلى حد ما بناءً على السمات الأنثروبولوجية، إلا أن البيانات الأخرى التي قد تسمح بتمثيل أكثر دقة تشمل لون العين ولون البشرة ولون الشعر وما إلى ذلك.
في حالة وجود كمية منخفضة من عينة الطب الشرعي أو عينة متدهورة، يمكن أن تكون طرق SNP بديلاً جيدًا لطرق المعاملات المشبوهة نظرًا لوفرة الواسمات المحتملة، وإمكانية التشغيل الآلي، واحتمال تقليل طول الجزء المطلوب إلى 60-80 نقطة أساس فقط.[28] في حالة عدم وجود تطابق STR في قاعدة بيانات ملفات تعريف الحمض النووي؛ يمكن استخدام أشكال تعدد الأشكال المختلفة للحصول على أدلة بخصوص العرق والنمط الظاهري والنسب وحتى الهوية.
علم الوراثة الدوائية[عدل]
ترتبط بعض أشكال النيوكلوتايد مع استقلاب الأدوية المختلفة.[29][30][31] يمكن أن تكون SNP طفرات، مثل الحذف، والتي يمكن أن تثبط أو تعزز النشاط الأنزيمي؛ يمكن أن يؤدي مثل هذا التغيير في النشاط الأنزيمي إلى انخفاض معدلات التمثيل الغذائي للدواء.[32] جمعية مجموعة واسعة من الأمراض التي تصيب الإنسان مثل السرطان، الأمراض المعدية (الإيدز، الجذام، التهاب الكبد، الخ) المناعة الذاتية، العصبية وأمراض أخرى كثيرة مع تعدد الأشكال المختلفة التي يمكن أن يتم حسب ذات الصلة علم الصيدلة الجيني أهداف لعلاج المخدرات.[33]
مرض[عدل]
قد يتسبب SNP الفردي في مرض مندلي، على الرغم من أنه بالنسبة للأمراض المعقدة، لا تعمل SNPs بشكل فردي، بدلاً من ذلك، فهي تعمل بالتنسيق مع SNPs الأخرى لإظهار حالة مرضية كما لوحظ في هشاشة العظام.[34] كان أحد أولى النجاحات في هذا المجال هو العثور على طفرة أساسية واحدة في المنطقة غير المشفرة من APOC3 (جين صميم البروتين الشحمي C3) الذي يرتبط بمخاطر أعلى للإصابة بارتفاع شحوم الدم وتصلب الشرايين.[35]
يمكن أن يكون لجميع أنواع النيوكلوتايد (SNPs) نمط ظاهري يمكن ملاحظته أو يمكن أن يؤدي إلى مرض:
- يمكن أن تظهر تعدد الأشكال في المناطق غير المشفرة في خطر أعلى للإصابة بالسرطان، [36] وقد تؤثر على بنية الرنا المرسال وقابلية الإصابة بالأمراض.[37] يمكن أن تغير SNPs غير المشفرة أيضًا مستوى التعبير عن الجين، مثل eQTL (التعبير عن موضع السمة الكمية).
- SNPs في مناطق الترميز:
- لا تؤدي البدائل المترادفة بحكم التعريف إلى تغيير الأحماض الأمينية في البروتين، ولكنها لا تزال تؤثر على وظيفتها بطرق أخرى. من الأمثلة على ذلك حدوث طفرة صامتة على ما يبدو في جين المقاومة للأدوية المتعددة 1 (MDR1)، والذي يرمز لمضخة غشاء خلوي تطرد الأدوية من الخلية، ويمكن أن تبطئ الترجمة وتسمح لسلسلة الببتيد بالانطواء لتشكل غير عادي تكون المضخة الطافرة أقل فاعلية (في بروتين MDR1 على سبيل المثال يغير تعدد الأشكال C1236T كودون GGC إلى GGT في موضع الأحماض الأمينية 412 من عديد الببتيد (كلاهما يشفر الجلايسين) ويغير تعدد الأشكال C3435T ATC إلى ATT في الموضع 1145 (كلاهما يشفر isoleucine)).[38]
- بدائل مجهولة:
- خطأ - يؤدي التغيير الفردي في القاعدة إلى تغيير في الأحماض الأمينية للبروتين وخللها الذي يؤدي إلى المرض (egc1580G> T SNP في جين LMNA - الموضع 1580 (nt) في تسلسل DNA (CGT codon) مما يؤدي إلى استبدال الجوانين مع الثايمين، ينتج كودون CTT في تسلسل الحمض النووي، ينتج على مستوى البروتين في استبدال الأرجينين بالليوسين في الموضع 527، [39] على مستوى النمط الظاهري، يتجلى هذا في تداخل خلل التنسج الفك السفلي ومتلازمة الشيخوخة المبكرة)
- طفرة هراء - نقطة تحول في تسلسل الحمض النووي الذي ينتج عنه سابق لأوانه كودون وقف، أو كودون هراء في كتب مرنا، وفي اقتطاع، غير مكتملة، والبروتين المنتج عادة لا يعمل (على سبيل المثال التليف الكيسي الناجم عن طفرة G542X في جين منظم توصيل غشاء التليف الكيسي).[40]
أمثلة[عدل]
- rs6311 وrs6313 هما SNPs في جين مستقبلات السيروتونين 5-HT2A على الكروموسوم البشري 13.[41]
- يسبب عامل SNP في جين F5 أهبة التخثر للعامل الخامس لايدن. [42]
- rs3091244 هو مثال على SNP التجريبي في جين CRP على الكروموسوم البشري 1.[43]
- رموز TAS2R38 لقدرة تذوق PTC، وتحتوي على 6 SNPs مشروحة.[44]
- rs148649884 و rs138055828 في ترميز الجين FCN1 M-ficolin شلّ قدرة ربط الترابط الخاص بـ M-ficolin المؤتلف.[45]
- يرتبط SNP intronic في جين إصلاح عدم تطابق الحمض النووي PMS2 (rs1059060، Ser775Asn) بزيادة تلف الحمض النووي للحيوانات المنوية وخطر الإصابة بالعقم عند الذكور.[46]
قواعد بيانات[عدل]
كما هو الحال بالنسبة للجينات، توجد قواعد بيانات المعلوماتية الحيوية لـ SNPs.
- قاعدة بيانات تعدد أشكال النكليوتيدات الفردية، هي قاعدة بيانات SNP من المركز الوطني لمعلومات التكنولوجيا الحيوية (NCBI). اعتبارًا من 8 يونيو 2015[تحديث] ، سجل dbSNP 149.735.377 تعدد الأشكال في البشر.[47][48]
- Kaviar [49] عبارة عن خلاصة وافية لـ SNPs من مصادر بيانات متعددة بما في ذلك dbSNP.
- SNPedia هي قاعدة بيانات على غرار الويكي تدعم شرح الجينوم الشخصي وتفسيره وتحليله.
- تصف قاعدة بيانات OMIM العلاقة بين تعدد الأشكال والأمراض (على سبيل المثال، تعطي الأمراض في شكل نصي)
- dbSAP - قاعدة بيانات أحادية لتعدد أشكال الأحماض الأمينية للكشف عن اختلاف البروتين [50]
- توفر قاعدة بيانات طفرة الجينات البشرية الطفرات الجينية التي تسبب أو ترتبط بأمراض وراثية للإنسان و SNPs الوظيفية
- مشروع HapMap الدولي، حيث يقوم الباحثون بتحديد علامة SNPs لتكون قادرة على تحديد مجموعة الأنماط الفردانية الموجودة في كل موضوع.
- يسمح GWAS Central للمستخدمين بالتحقيق بصريًا في بيانات الارتباط الفعلية على مستوى الملخص في دراسة أو أكثر من دراسات الارتباط على مستوى الجينوم.
قامت مجموعة عمل خريطة SNP الدولية بتعيين التسلسل الذي يحيط بكل SNP من خلال المحاذاة مع التسلسل الجيني للنسخ كبيرة الإدراج في Genebank. تم تحويل هذه المحاذاة إلى إحداثيات كروموسومية كما هو موضح في الجدول 1.[51] زادت هذه القائمة بشكل كبير منذ ذلك الحين، على سبيل المثال، تسرد قاعدة بيانات Kaviar الآن 162 مليون متغير للنيوكليوتيدات المفردة (SNVs).
| كروموسوم | الطول (bp) | تعدد أشكال النوكليوتيدات المفردة | تعدد أشكال النوكليوتيدات المفردة SNPs | ||
|---|---|---|---|---|---|
| إجمالي SNPs | kb per SNP | إجمالي SNPs | kb per SNP | ||
| 1 | 214,066,000 | 129,931 | 1.65 | 75,166 | 2.85 |
| 2 | 222,889,000 | 103,664 | 2.15 | 76,985 | 2.90 |
| 3 | 186,938,000 | 93,140 | 2.01 | 63,669 | 2.94 |
| 4 | 169,035,000 | 84,426 | 2.00 | 65,719 | 2.57 |
| 5 | 170,954,000 | 117,882 | 1.45 | 63,545 | 2.69 |
| 6 | 165,022,000 | 96,317 | 1.71 | 53,797 | 3.07 |
| 7 | 149,414,000 | 71,752 | 2.08 | 42,327 | 3.53 |
| 8 | 125,148,000 | 57,834 | 2.16 | 42,653 | 2.93 |
| 9 | 107,440,000 | 62,013 | 1.73 | 43,020 | 2.50 |
| 10 | 127,894,000 | 61,298 | 2.09 | 42,466 | 3.01 |
| 11 | 129,193,000 | 84,663 | 1.53 | 47,621 | 2.71 |
| 12 | 125,198,000 | 59,245 | 2.11 | 38,136 | 3.28 |
| 13 | 93,711,000 | 53,093 | 1.77 | 35,745 | 2.62 |
| 14 | 89,344,000 | 44,112 | 2.03 | 29,746 | 3.00 |
| 15 | 73,467,000 | 37,814 | 1.94 | 26,524 | 2.77 |
| 16 | 74,037,000 | 38,735 | 1.91 | 23,328 | 3.17 |
| 17 | 73,367,000 | 34,621 | 2.12 | 19,396 | 3.78 |
| 18 | 73,078,000 | 45,135 | 1.62 | 27,028 | 2.70 |
| 19 | 56,044,000 | 25,676 | 2.18 | 11,185 | 5.01 |
| 20 | 63,317,000 | 29,478 | 2.15 | 17,051 | 3.71 |
| 21 | 33,824,000 | 20,916 | 1.62 | 9,103 | 3.72 |
| 22 | 33,786,000 | 28,410 | 1.19 | 11,056 | 3.06 |
| X | 131,245,000 | 34,842 | 3.77 | 20,400 | 6.43 |
| Y | 21,753,000 | 4,193 | 5.19 | 1,784 | 12.19 |
| مرجع | 15,696,674 | 14,534 | 1.08 | ||
| الإجمالي | 2,710,164,000 | 1,419,190 | 1.91 | 887,450 | 3.05 |
التسمية[عدل]

يمكن أن تكون التسمية الخاصة بـ SNPs مربكة: يمكن أن توجد العديد من الاختلافات لـ SNP الفردي، ولم يتم تحقيق الإجماع بعد.
معيار rs ### هو الذي تم تبنيه من قبل قاعدة بيانات تعدد أشكال النكليوتيدات الفردية (dbSNP) ويستخدم البادئة "rs"، لـ «مرجع SNP»، متبوعًا برقم فريد وتعسفي.[52] غالبًا ما يشار إلى SNPs برقم dbSNP rs الخاص بهم، كما في الأمثلة أعلاه.
تستخدم جمعية تباين الجينوم البشري (HGVS) معيارًا ينقل المزيد من المعلومات حول SNP. الأمثلة هي:
- c.76A>T: لمنطقة التشفير"c"، متبوعًا برقم لموضع النيوكليوتيدات، متبوعًا باختصار من حرف واحد للنيوكليوتيدات (A، C، G، T أو U)، متبوعًا بعلامة أكبر من (">") للإشارة استبدال، متبوعًا باختصار النيوكليوتيد الذي يحل محل السابق
- P": Ser123Arg.P" للبروتين، متبوعًا باختصار مكون من ثلاثة أحرف للحمض الأميني، متبوعًا برقم لموضع الحمض الأميني، متبوعًا باختصار الحمض الأميني الذي يحل محل الأول.
تحليل SNP[عدل]
عادة ما تكون SNPs ثنائية النواة وبالتالي يسهل فحصها.[51] تتضمن الطرق التحليلية لاكتشاف أشكال تعدد الأشكال الجديدة واكتشاف أشكال تعدد الأشكال المعروفة:
- تسلسل الحمض النووي.[53]
- الكهربائي الشعري.[54]
- مطياف الكتلة.[55]
- تعدد أشكال التشكل أحادي الخيط (SSCP).[56]
- تمديد قاعدة واحدة.
- التحليل الكهروكيميائي.
- تغيير طبيعة HPLC والهلام الكهربائي.
- تقييد طول جزء تعدد الأشكال.
- تحليل التهجين.
برامج للتنبؤ بتأثيرات SNP[عدل]
مجموعة مهمة من SNPs هي تلك التي تتوافق مع الطفرات الخاطئة التي تسبب تغير الأحماض الأمينية على مستوى البروتين. يمكن أن يكون للطفرة النقطية لبقايا معينة تأثير مختلف على وظيفة البروتين (من عدم وجود تأثير إلى تعطيل وظيفته بالكامل). عادةً ما يكون للتغيير في الأحماض الأمينية ذات الحجم المماثل والخصائص الفيزيائية الكيميائية (مثل الاستبدال من الليوسين إلى الفالين) تأثير خفيف وعكس ذلك. وبالمثل، إذا عطلت SNP عناصر البنية الثانوية (مثل استبدال البرولين في منطقة ألفا الحلزونية)، فقد تؤثر هذه الطفرة عادةً على بنية البروتين ووظيفته بالكامل. باستخدام تلك القواعد البسيطة والعديد من القواعد المشتقة الأخرى للتعلم الآلي، تم تطوير مجموعة من البرامج للتنبؤ بتأثير SNP:
- SIFT يوفر هذا البرنامج نظرة ثاقبة حول كيفية تأثير المختبر على خطأ أو طفرة غير مترادفة على وظيفة البروتين بناءً على الخصائص الفيزيائية للحمض الأميني وتماثل التسلسل.
- تقدر LIST [57][58] (الهوية المحلية والأصناف المشتركة) الضرر المحتمل للطفرات الناتجة عن تغيير وظائف البروتين. ويستند إلى افتراض أن الاختلافات التي لوحظت في الأنواع وثيقة الصلة تكون أكثر أهمية عند تقييم الحفظ مقارنة بتلك الموجودة في الأنواع ذات الصلة البعيدة.
- SNAP2.
- مشتبه فيه.
- بوليفين -2.
- توقع SNP .
- ميوتيشن تيستر: الموقع الرسمي
- متنبئ التأثير المتغير من مشروع Ensembl
- SNPViz [59] يوفر هذا البرنامج تمثيلًا ثلاثي الأبعاد للبروتين المصاب، مع تسليط الضوء على تغير الأحماض الأمينية حتى يتمكن الأطباء من تحديد الإمراضية للبروتين الطافرة.
- إثبات
- PhyreRisk هي قاعدة بيانات تقوم بتعيين المتغيرات لهياكل البروتين التجريبية والمتوقعة.[60]
- Missense3D هي أداة تقدم تقريرًا كيميائيًا مجسمًا عن تأثير متغيرات الخطأ على بنية البروتين.[61]
انظر أيضًا[عدل]
- أفيميتريكس.
- هاب ماب.
- إلومينا.
- مشروع هاب ماب الدولي.
- تكرار ترادفي قصير (STR).
- تمديد قاعدة واحدة.
- مجموعة SNP.
- التنميط الجيني SNP.
- SNPedia.
- Snpstr.
- اتصال SNV من بيانات NGS.
- علامة SNP.
- تاكمان.
- فيريوم (متغير).
المراجع[عدل]
- ^ Barreiro LB؛ Laval G؛ Quach H؛ Patin E؛ Quintana-Murci L. (2008). "Natural selection has driven population differentiation in modern humans". Nature Genetics. ج. 40 ع. 3: 340–345. DOI:10.1038/ng.78. PMID:18246066.
- ^ Nachman, Michael W. (2001). "Single nucleotide polymorphisms and recombination rate in humans". Trends in genetics. ج. 17 ع. 9: 481–485. DOI:10.1016/S0168-9525(01)02409-X. PMID:11525814.
- ^ "single-nucleotide polymorphism / SNP | Learn Science at Scitable". www.nature.com. مؤرشف من الأصل في 2015-11-10. اطلع عليه بتاريخ 2015-11-13.
- ^ "A specific chemical difference between the globins of normal human and sickle-cell anaemia haemoglobin". Nature. ج. 178 ع. 4537: 792–4. أكتوبر 1956. Bibcode:1956Natur.178..792I. DOI:10.1038/178792a0. PMID:13369537.
- ^ "beta 0 thalassemia, a nonsense mutation in man". Proceedings of the National Academy of Sciences of the United States of America. ج. 76 ع. 6: 2886–9. يونيو 1979. Bibcode:1979PNAS...76.2886C. DOI:10.1073/pnas.76.6.2886. PMC:383714. PMID:88735.
- ^ "Cystic fibrosis patients bearing both the common missense mutation Gly----Asp at codon 551 and the delta F508 mutation are clinically indistinguishable from delta F508 homozygotes, except for decreased risk of meconium ileus". American Journal of Human Genetics. ج. 51 ع. 2: 245–50. أغسطس 1992. PMC:1682672. PMID:1379413.
- ^ "APOE and neuroenergetics: an emerging paradigm in Alzheimer's disease". Neurobiology of Aging. ج. 34 ع. 4: 1007–17. أبريل 2013. DOI:10.1016/j.neurobiolaging.2012.10.011. PMC:3545040. PMID:23159550.
- ^ http://resources.qiagenbioinformatics.com/manuals/clcgenomicsworkbench/700/Variant_types.html#:~:text=SNV%20is%20preferred%20over%20SNP,or%20more%20SNVs%20in%20succession. نسخة محفوظة 2020-07-16 على موقع واي باك مشين.
- ^ ISO - ISO 20395:2019 - Biotechnology — Requirements for evaluating the performance of quantification methods for nucleic acid target sequences — qPCR and dPCR نسخة محفوظة 2020-11-01 على موقع واي باك مشين.
- ^ "Haplotype block partitioning and tag SNP selection using genotype data and their applications to association studies". Genome Research. ج. 14 ع. 5: 908–16. مايو 2004. DOI:10.1101/gr.1837404. PMC:479119. PMID:15078859.
- ^ "Single nucleotide polymorphisms: a new paradigm for molecular marker technology and DNA polymorphism detection with emphasis on their use in plants". Current Science. ج. 80 ع. 4: 524–535. 25 فبراير 2001. مؤرشف من الأصل في 2017-02-13.
- ^ "A global reference for human genetic variation". Nature. ج. 526 ع. 7571: 68–74. أكتوبر 2015. Bibcode:2015Natur.526...68T. DOI:10.1038/nature15393. PMC:4750478. PMID:26432245.
- ^ "Natural selection has driven population differentiation in modern humans". Nature Genetics. ج. 40 ع. 3: 340–5. مارس 2008. DOI:10.1038/ng.78. PMID:18246066.
- ^ "Single nucleotide polymorphisms and recombination rate in humans". Trends in Genetics. ج. 17 ع. 9: 481–5. سبتمبر 2001. DOI:10.1016/S0168-9525(01)02409-X. PMID:11525814.
- ^ "Heterogeneous distribution of SNPs in the human genome: microsatellites as predictors of nucleotide diversity and divergence". Genomics. ج. 95 ع. 3: 151–9. مارس 2010. DOI:10.1016/j.ygeno.2009.12.003. PMID:20026267.
- ^ "Enrichment of Minor Alleles of Common SNPs and Improved Risk Prediction for Parkinson's Disease". PLOS ONE. ج. 10 ع. 7: e0133421. 24 يوليو 2015. Bibcode:2015PLoSO..1033421Z. DOI:10.1371/journal.pone.0133421. PMC:4514478. PMID:26207627.
{{استشهاد بدورية محكمة}}: صيانة الاستشهاد: دوي مجاني غير معلم (link) - ^ Hivert، Valentin؛ Leblois، Raphaël؛ Petit، Eric J.؛ Gautier، Mathieu؛ Vitalis، Renaud (30 يوليو 2018). "Measuring Genetic Differentiation from Pool-seq Data". Genetics. ج. 210 ع. 1: 315–330. DOI:10.1534/genetics.118.300900. ISSN:0016-6731. مؤرشف من الأصل في 2020-12-18.
- ^ Ekblom، R؛ Galindo، J (8 ديسمبر 2010). "Applications of next generation sequencing in molecular ecology of non-model organisms". Heredity. ج. 107 ع. 1: 1–15. DOI:10.1038/hdy.2010.152. ISSN:0018-067X. مؤرشف من الأصل في 2020-05-28.
- ^ Ellegren، Hans (يناير 2014). "Genome sequencing and population genomics in non-model organisms". Trends in Ecology & Evolution. ج. 29 ع. 1: 51–63. DOI:10.1016/j.tree.2013.09.008. ISSN:0169-5347. مؤرشف من الأصل في 2020-10-10.
- ^ Dorant, Yann; Benestan, Laura; Rougemont, Quentin; Normandeau, Eric; Boyle, Brian; Rochette, Rémy; Bernatchez, Louis (2019). "Comparing Pool-seq, Rapture, and GBS genotyping for inferring weak population structure: The American lobster (Homarus americanus) as a case study". Ecology and Evolution (بالإنجليزية). 9 (11): 6606–6623. DOI:10.1002/ece3.5240. ISSN:2045-7758. PMC:6580275. PMID:31236247. Archived from the original on 2020-02-12.
- ^ Vendrami، David L. J.؛ Telesca، Luca؛ Weigand، Hannah؛ Weiss، Martina؛ Fawcett، Katie؛ Lehman، Katrin؛ Clark، M. S.؛ Leese، Florian؛ McMinn، Carrie. "RAD sequencing resolves fine-scale population structure in a benthic invertebrate: implications for understanding phenotypic plasticity". Royal Society Open Science. ج. 4 ع. 2: 160548. DOI:10.1098/rsos.160548. PMC:5367306. PMID:28386419. مؤرشف من الأصل في 2020-05-27.
- ^ Carlson، Bruce (15 يونيو 2008). "SNPs — A Shortcut to Personalized Medicine". ماري آن ليبيرت. ج. 28 ع. 12. مؤرشف من الأصل في 2010-12-26. اطلع عليه بتاريخ 2008-07-06.
(subtitle) Medical applications are where the market's growth is expected
- ^ "Application of a high-resolution genetic map for chromosome-scale genome assembly and fine QTLs mapping of seed size and weight traits in castor bean". Scientific Reports. ج. 9 ع. 1: 11950. أغسطس 2019. Bibcode:2019NatSR...911950Y. DOI:10.1038/s41598-019-48492-8. PMC:6697702. PMID:31420567.
{{استشهاد بدورية محكمة}}: الوسيط|إظهار المؤلفين=6غير صالح (مساعدة) - ^ "Challenges in the association of human single nucleotide polymorphism mentions with unique database identifiers". BMC Bioinformatics. 12 Suppl 4: S4. 2011. DOI:10.1186/1471-2105-12-S4-S4. PMC:3194196. PMID:21992066.
{{استشهاد بدورية محكمة}}: صيانة الاستشهاد: دوي مجاني غير معلم (link) - ^ Butler، John M. (2010). Fundamentals of forensic DNA typing. Burlington, MA: Elsevier/Academic Press. ISBN:9780080961767.
- ^ "Forensic SNP Genotyping using Nanopore MinION Sequencing". Scientific Reports. ج. 7: 41759. فبراير 2017. Bibcode:2017NatSR...741759C. DOI:10.1038/srep41759. PMC:5290523. PMID:28155888.
- ^ "Developing a SNP panel for forensic identification of individuals". Forensic Science International. ج. 164 ع. 1: 20–32. ديسمبر 2006. DOI:10.1016/j.forsciint.2005.11.017. PMID:16360294.
- ^ "Forensically relevant SNP classes". BioTechniques. ج. 44 ع. 5: 603–8, 610. أبريل 2008. DOI:10.2144/000112806. PMID:18474034. مؤرشف من الأصل في 2011-04-13.
- ^ "Clinical relevance of genetic polymorphisms in the human CYP2C subfamily". British Journal of Clinical Pharmacology. ج. 52 ع. 4: 349–55. أكتوبر 2001. DOI:10.1046/j.0306-5251.2001.01499.x. PMC:2014584. PMID:11678778.
- ^ "CYP2C9 genotype as a predictor of drug disposition in humans". Methods and Findings in Experimental and Clinical Pharmacology. ج. 26 ع. 6: 463–72. يوليو–أغسطس 2004. PMID:15349140.
- ^ "Functional SNPs of the breast cancer resistance protein-therapeutic effects and inhibitor development". Cancer Letters. ج. 234 ع. 1: 73–80. مارس 2006. DOI:10.1016/j.canlet.2005.04.039. PMID:16303243.
- ^ Butler، Merlin G. (2018). "Pharmacogenetics and Psychiatric Care: A Review and Commentary". Journal of Mental Health & Clinical Psychology. ج. 2 ع. 2: 17–24. DOI:10.29245/2578-2959/2018/2.1120. PMC:6291002. PMID:30556062.
- ^ "Single-nucleotide polymorphism in genome-wide association of human population: A tool for broad spectrum service". Egyptian Journal of Medical Human Genetics. ج. 14 ع. 2: 123–134. أبريل 2013. DOI:10.1016/j.ejmhg.2012.08.001.
- ^ "SNP-SNP interactions within APOE gene influence plasma lipids in postmenopausal osteoporosis". Rheumatology International. ج. 31 ع. 3: 421–3. مارس 2011. DOI:10.1007/s00296-010-1449-7. PMID:20340021.
- ^ "DNA polymorphism adjacent to human apoprotein A-1 gene: relation to hypertriglyceridaemia". Lancet. ج. 1 ع. 8322: 444–6. فبراير 1983. DOI:10.1016/S0140-6736(83)91440-X. PMID:6131168.
- ^ "Regulatory Variants and Disease: The E-Cadherin -160C/A SNP as an Example". Molecular Biology International. ج. 2014: 967565. 2014. DOI:10.1155/2014/967565. PMC:4167656. PMID:25276428.
{{استشهاد بدورية محكمة}}: صيانة الاستشهاد: دوي مجاني غير معلم (link) - ^ "IFNL3 mRNA structure is remodeled by a functional non-coding polymorphism associated with hepatitis C virus clearance". Scientific Reports. ج. 5: 16037. نوفمبر 2015. Bibcode:2015NatSR...516037L. DOI:10.1038/srep16037. PMC:4631997. PMID:26531896.
- ^ "A "silent" polymorphism in the MDR1 gene changes substrate specificity". Science. ج. 315 ع. 5811: 525–8. يناير 2007. Bibcode:2007Sci...315..525K. DOI:10.1126/science.1135308. PMID:17185560. مؤرشف من الأصل في 2020-08-07.
- ^ "A novel homozygous p.Arg527Leu LMNA mutation in two unrelated Egyptian families causes overlapping mandibuloacral dysplasia and progeria syndrome". European Journal of Human Genetics. ج. 20 ع. 11: 1134–40. نوفمبر 2012. DOI:10.1038/ejhg.2012.77. PMC:3476705. PMID:22549407.
- ^ "CFTR mutation analysis and haplotype associations in CF patients". Molecular Genetics and Metabolism. ج. 105 ع. 2: 249–54. فبراير 2012. DOI:10.1016/j.ymgme.2011.10.013. PMC:3551260. PMID:22137130.
- ^ "Anger- and aggression-related traits are associated with polymorphisms in the 5-HT-2A gene". Journal of Affective Disorders. ج. 96 ع. 1–2: 75–81. نوفمبر 2006. DOI:10.1016/j.jad.2006.05.016. PMID:16814396.
- ^ "Factor V Leiden thrombophilia". Genetics in Medicine. ج. 13 ع. 1: 1–16. يناير 2011. DOI:10.1097/GIM.0b013e3181faa0f2. PMID:21116184.
- ^ "Genotyping of triallelic SNPs using TaqMan PCR". Molecular and Cellular Probes. ج. 21 ع. 3: 171–6. يونيو 2007. DOI:10.1016/j.mcp.2006.10.005. PMID:17161935.
- ^ "Bitter taste study in a sardinian genetic isolate supports the association of phenylthiocarbamide sensitivity to the TAS2R38 bitter receptor gene". Chemical Senses. ج. 29 ع. 8: 697–702. أكتوبر 2004. DOI:10.1093/chemse/bjh074. PMID:15466815.
- ^ "Non-synonymous polymorphisms in the FCN1 gene determine ligand-binding ability and serum levels of M-ficolin". PLOS ONE. ج. 7 ع. 11: e50585. 28 نوفمبر 2012. Bibcode:2012PLoSO...750585A. DOI:10.1371/journal.pone.0050585. PMC:3509001. PMID:23209787.
{{استشهاد بدورية محكمة}}: صيانة الاستشهاد: دوي مجاني غير معلم (link) - ^ "Common variants in mismatch repair genes associated with increased risk of sperm DNA damage and male infertility". BMC Medicine. ج. 10: 49. مايو 2012. DOI:10.1186/1741-7015-10-49. PMC:3378460. PMID:22594646.
{{استشهاد بدورية محكمة}}: صيانة الاستشهاد: دوي مجاني غير معلم (link) - ^ National Center for Biotechnology Information, United States National Library of Medicine. 2014. NCBI dbSNP build 142 for human. "[DBSNP-announce] DBSNP Human Build 142 (GRCh38 and GRCh37.p13)". مؤرشف من الأصل في 2017-09-10. اطلع عليه بتاريخ 2017-09-11.
- ^ National Center for Biotechnology Information, United States National Library of Medicine. 2015. NCBI dbSNP build 144 for human. Summary Page. "DBSNP Summary". مؤرشف من الأصل في 2017-09-10. اطلع عليه بتاريخ 2017-09-11.
- ^ "Kaviar: an accessible system for testing SNV novelty". Bioinformatics. ج. 27 ع. 22: 3216–7. نوفمبر 2011. DOI:10.1093/bioinformatics/btr540. PMC:3208392. PMID:21965822.
- ^ "dbSAP: single amino-acid polymorphism database for protein variation detection". Nucleic Acids Research. ج. 45 ع. D1: D827–D832. يناير 2017. DOI:10.1093/nar/gkw1096. PMC:5210569. PMID:27903894.
- ^ أ ب "A map of human genome sequence variation containing 1.42 million single nucleotide polymorphisms". Nature. ج. 409 ع. 6822: 928–33. فبراير 2001. Bibcode:2001Natur.409..928S. DOI:10.1038/35057149. PMID:11237013.
- ^ "Clustered RefSNPs (rs) and Other Data Computed in House". SNP FAQ Archive. Bethesda (MD): U.S. National Center for Biotechnology Information. 2005.
- ^ "An SNP map of the human genome generated by reduced representation shotgun sequencing". Nature. ج. 407 ع. 6803: 513–6. سبتمبر 2000. Bibcode:2000Natur.407..513A. DOI:10.1038/35035083. PMID:11029002.
- ^ "Identification of base pairs in single-nucleotide polymorphisms by MutS protein-mediated capillary electrophoresis". Analytical Chemistry. ج. 78 ع. 6: 2035–8. مارس 2006. DOI:10.1021/ac0520386. PMID:16536443.
- ^ "Genetic identification by mass spectrometric analysis of single-nucleotide polymorphisms: ternary encoding of genotypes". Analytical Chemistry. ج. 72 ع. 14: 3298–302. يوليو 2000. DOI:10.1021/ac991390e. PMID:10939403.
- ^ "Estimation of SNP allele frequencies by SSCP analysis of pooled DNA". Single Nucleotide Polymorphisms. Methods in Molecular Biology. ج. 578. 2009. ص. 193–207. DOI:10.1007/978-1-60327-411-1_12. ISBN:978-1-60327-410-4. PMID:19768595.
- ^ "Improved measures for evolutionary conservation that exploit taxonomy distances". Nature Communications. ج. 10 ع. 1: 1556. أبريل 2019. Bibcode:2019NatCo..10.1556M. DOI:10.1038/s41467-019-09583-2. PMC:6450959. PMID:30952844.
- ^ Nawar Malhis؛ Matthew Jacobson؛ Steven J. M. Jones؛ Jörg Gsponer (2020). "LIST-S2: Taxonomy Based Sorting of Deleterious Missense Mutations Across Species". Nucleic Acids Research. ج. 48 ع. W1: W154–W161. DOI:10.1093/nar/gkaa288. PMC:7319545. PMID:32352516.
- ^ "View of SNPViz - Visualization of SNPs in proteins". genomicscomputbiol.org (بالإنجليزية الأمريكية). Archived from the original on 2020-08-07. Retrieved 2018-10-20.
- ^ "PhyreRisk: A Dynamic Web Application to Bridge Genomics, Proteomics and 3D Structural Data to Guide Interpretation of Human Genetic Variants". Journal of Molecular Biology. ج. 431 ع. 13: 2460–2466. يونيو 2019. DOI:10.1016/j.jmb.2019.04.043. PMC:6597944. PMID:31075275.
{{استشهاد بدورية محكمة}}: الوسيط|إظهار المؤلفين=6غير صالح (مساعدة) - ^ "Can Predicted Protein 3D Structures Provide Reliable Insights into whether Missense Variants Are Disease Associated?". Journal of Molecular Biology. ج. 431 ع. 11: 2197–2212. مايو 2019. DOI:10.1016/j.jmb.2019.04.009. PMC:6544567. PMID:30995449.
قراءة متعمقة[عدل]
==
- "Glossary". Nature Reviews. مؤرشف من الأصل في 2016-03-05.
- Human Genome Project Information — SNP Fact Sheet
روابط خارجية ==
- موارد NCBI - مقدمة إلى SNPs من NCBI
- SNP Consortium LTD - بحث SNP
- قاعدة بيانات NCBI dbSNP - «مستودع مركزي لكل من بدائل النوكليوتيدات ذات القاعدة الفردية والحذف القصير وتعدد الأشكال»
- HGMD - قاعدة بيانات طفرة الجينات البشرية، تتضمن طفرات نادرة وتعدد الأشكال الوظيفية
- GWAS Central - قاعدة بيانات مركزية لنتائج الارتباط الجيني على مستوى الملخص
- مشروع 1000 جينوم - فهرس عميق للتنوع الجيني البشري
- WatCut - أداة عبر الإنترنت لتصميم فحوصات SNP-RFLP
- SNPStats - SNPStats، أداة ويب لتحليل دراسات الارتباط الجيني
- تقييد الصفحة الرئيسية - مجموعة من الأدوات لتقييد الحمض النووي واكتشاف SNP، بما في ذلك تصميم بادئات مطفرة
- الرابطة الأمريكية لأبحاث السرطان ورقة حقائق مفاهيم مفاهيم السرطان عن تعدد الأشكال
- PharmGKB - قاعدة معارف علم الوراثة الدوائية وعلم الوراثة الصيدلانية، وهي مورد لأشكال النيوكلوتايد المرتبطة باستجابة الأدوية ونتائج المرض.
- GEN-SNiP - أداة على الإنترنت تحدد تعدد الأشكال في اختبار تسلسل الحمض النووي.
- قواعد تسمية الجينات، والعلامات الجينية، والأليل، والطفرات في الفئران والجرذان
- إرشادات HGNC لتسميات الجينات البشرية
- توقع تأثير SNP مع تكامل المجرة
- افتح SNP - بوابة لمشاركة نتائج اختبار SNP الخاصة بك
- dbSAP - قاعدة بيانات SNP للكشف عن تباين البروتين
| في كومنز صور وملفات عن: تعدد أشكال النوكليوتيدات المفردة |
