علم التخلق
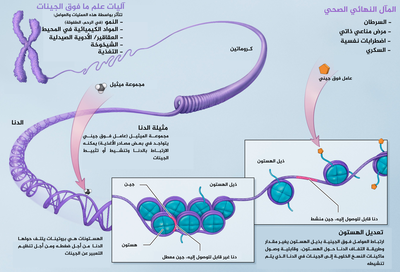
علم ما فوق الجينات (بالإنجليزية: Epigenetics) أو علمُ التَّخَلُّق[1] أو علم التخلق المتعاقب أو علم التخلق المتوالي[2] هو دراسة التغيرات التي تحدث لنمط ظاهري وراثي لأسباب لا تتعلق بتغير تسلسل الدنا.[3] والسابقة الإغريقية "epi" (ἐπι «فوق، خارج، حول») تعني في علم ما فوق الجينات ميزات أو آليات تكون «فضلا على» أو «زيادة عن» الأسس الجينية التقليدية للوراثة.[4] لعلم ما فوق الجينات عادة دور في التغيُّرات التي تؤثر على نشاط الجين والتعبير عنه،[ملاحظة 1] لكن يمكن استخدام المصطلح كذلك لوصف أي تغيرٍ وراثي في نمط ظاهري. يمكن أن تَنتج هذه التأثيرات في السمات الظاهرية الفيسيولوجية والخلوية من عوامل خارجية أو بيئية، أو تكون جزءا من النمو الطبيعي. التعريف النموذجي لعلم ما فوق الجينات يتطلب أن تكون هذه التغيرات في الأنماط الظاهرية وراثيةً[7][8] في نسل كلٍ من الخلايا أو الكائنات.
المصطلح يشير كذلك إلى التغيرات في حد ذاتها: التغيُّرات الوظيفية للجينوم التي لا تشمل تغيرات في تسلسل النوكليوتيدات. من الأمثلة على الآليات التي تُحدث مثل هذه التغيرات: مثيلة الدنا وتعديل الهستونات اللذان يغير كل منهما كيفية التعبير عن الجينات من دون تغيير تسلسل الدنا الخاص بها. يمكن التحكم في التعبير الجيني عبر البروتينات المثبطة التي ترتبط بمناطق الكاتم في الدنا. يمكن أن تستمر هذه التغيرات فوق الجينية خلال انقسامات الخلية طوال مدة حياة الخلية، ويمكن أن تستمر حتى لعدة أجيال. ورغم أن هذه التغيرات لا تشمل تغيرات في تسلسل الدنا الخاص بالكائن،[9] إلا أن العوامل فوق الجينية تتسبب في تصرف الجينات (التعبير عن نفسها) على نحو مختلف.[10]
أحد الأمثلة على تغيرٍ فوق جينيٍ في علم أحياء حقيقيات النوى هو عملية التمايز الخلوي. أثناء التخلق التشكلي تصبح الخلايا الجذعية شاملة القدرة مختلف السلالات الخلوية متعددة القدرات التي تكون الجنين، والتي تصبح بدورها خلايا متمايزة كليا. بعبارة أخرى تنقسم بويضة ملقحة واحدة -الزيجوت- وتستمر بالانقسام حتى تنتج جميع أنواع الخلايا الخاصة بالكائن مثل: العصبونات، الخلايا العضلية، النسيج الطلائي والبطانة الغشائية والأوعية الدموية... عبر تنشيط جينات معينة وتثبيط التعبير عن أخرى.[11]
تاريخيا، تم وصف بعض الظواهر التي ليست وراثية بالضرورة على أنها فوق جينية. على سبيل المثال اُستخدم المصطلح «فوق جيني» لوصف أي تعديلٍ لمناطق صبغية، خاصة تعديلات الهستون سواء كانت هذه التعديلات وراثية أم لم تكن وكذلك سواء كانت مرتبطة بنمط ظاهري أو لا. التعريف المجمع عليه حاليا يتطلب أن تكون السمة وراثيةً ليتم اعتبارها بأنها فوق جينية.[8]
تاريخ وتعريفات
[عدل]ظهر مصطلح علم الجينات الفوقي باستخدامه المعاصر في عقد 1990، لكن ولعدة سنوات كان يُستخدم بمعانٍ مختلفة قليلا.[12] أُجْمِعَ على صياغة تعريف لمفهوم سمة فوق جينية بأنها «نمط ظاهري وراثي مستقر ينتج عن تغيراتٍ في صبغي من دون تغيرات في تسلسل الدنا» في اجتماع بمختبر كولد سبرينغ هاربر في2008،[8] ورغم ذلك مازالت تُستخدم تعريفات بديلة تشمل السمات غير الوراثية.[13]
مصطلح تخلق فوقي (epigenesis) يعني بشكل عام «نمو إضافي» وهو يستخدم في اللغة الإنجليزية منذ القرن السابع عشر.[14]
استقناء وادينجتون، عقد 1940
[عدل]من المعنى العام لكلمة تخلق فوقي (epigenesis) - والصفة ذات الصلة «فوق جيني» (epigenetic) - ابتكر عالم الأجنة البريطاني كونراد وادينجتون مصطلح علم مافوق الجينات (epigenetics) سنة 1942 قياسا على مصطلح علم الجينات الظاهري (phenogenetics) الذي صاغه فالنتين هيكر.[15] يشير علم الجينات الفوقي في سياق البيولوجيا في مرحلة النمو الجنيني إلى تمايز الخلايا الجذعية من حالتها الأولية شاملة القدرة.[16]
حين صاغ وادينجتون المصطلح، كانت الطبيعة المادية للجينات ودورها في الوراثة غير معروف. واستخدمها بدل ذلك كنموذج مفاهيمي لكيفية احتمال تآثر المكونات الجينية مع المحيط لإنتاج نمط ظاهري، واستخدم عبارة «مشهد عام فوق جيني» كتعبير مجازي حول النمو البيولوجي. آمن وادينجتون أن مصائر الخلية تُحدد أثناء النمو في عملية سماها الاستقناء تماما مثل تدحرج كجة إلى أسفل نقطة من مرتفع محلي.[17] واقترح وادينجتون مشاهدة تمايز خلوي متزايد غير قابل للعكس مثل ارتفاع التلال بين الوديان التي تسافر الكجج (شبهها بالخلايا) فيها.[18]
في الوقت الحالي، تم ترسيم فكرة وادينجتون المتعلقة «بالمشهد العام فوق الجيني» بصرامة في سياق منهج ديناميكيات النظام لدراسة مصير الخلية.[19][20] يُتوقع أن تحديد مصير الخلية يُظهر بعض الديناميكيات مثل جاذب-تقارب (يمكن أن يكون الجاذب نقطة توازن، دورة الحد أو جاذب غريب) أو تذبذبي.[20]
التعريف المعاصر
[عدل]يعرف روبن هوليداي علم الجينات الفوقي على أنه «العلم الذي يدرس آليات التحكم الزماني والمكاني في نشاط الجين أثناء نمو الكائنات المعقدة».[21] وبالتالي، في معنى أكثر شمولا: يمكن أن يُستخدم مصطلح فوق جيني لوصف أي شيء يؤثر على نمو كائنٍ باستثناء تسلسل الدنا. تتّبع الاستخداماتُ الأحدث للمصطلح تعريفاتٍ أكثر صرامة، وقد عرفه أرثر ريغز وزملاؤه على أنه «العلم الذي يدرس تغيرات الانقسام المتساو و/أو الانقسام المنصف الوراثية في وظيفة جين والتي لا يمكن تفسيرها بتغيرات في تسلسل الدنا».[22]
يُستخدم المصطلح كذلك لوصف العمليات التي لم يُثبت أنها وراثية مثل بعض أنواع تعديل الهستون، وبالتالي هناك محاولات لإعادة تعريف «علم الجينات الفوقي» بشكل أوسع يسمح بتجنب القيود التي تتطلب الوروثية. على سبيل المثال يُعرف أدريان بيرد علم الجينات الفوقي بأنه «تكيف بنيوي لمناطق صبغية لكي تسجل أو تشير أو تخلِّد حالات نشاط معدَّلة».[9] هذا التعريف سيكون شاملا للتعديلات المؤقتة المتعلقة بترميم الدنا أو أطوار دورة الخلية وكذلك التغييرات المستقرة المحافظ عليها خلال عدة أجيال خلوية، لكنه سيستبعد عمليات أخرى مثل قولبة معمارية الغشاء والبريونات إلا إذا كان لها تأثير على وظيفة الصبغي. إعادت تعريف كهذه ليست مقبولة بالإجماع ومازالت محل نقاشات.[7] يستخدم «مشروع خريطة طريق علم الجينات الفوقي» الخاص بمعاهد الصحة الوطنية الأمريكية والجاري منذ 2016 التعريف التالي: «لأهداف هذا المشروع، يشير علم الجينات الفوقي إلى كِلا التغيرات الوراثية في نشاط الجين والتعبير عنه (في نسل الخلايا أو الأفراد)، وكذلك التغيرات المستقرة طويلة الأمد في قدرة النسخ في الخلية والتي ليست وراثية بالضرورة.»[13] في 2008، تم الإجماع على تعريف سمة فوق جينية على أنها «نمط جيني مستقر وراثي ناتج عن تغيرات في الصبغي من دون تغيرات في تسلسل الدنا»، في اجتماع بمختبر كولد سبرينغ هاربر.[8]
ولَّد تشابه المصطلح علم الجينات الفوقي بمصطلح علم الجينات العديد من الاستخدامات الموازية. مصطلح «جينوم فوقي» استخدام موازي لمصطلح جينوم ويشير إلى الحالة الإجمالية فوق الجينية للخلية. وعلم الجينوم الفوقي يشير إلى مجمل دراسات التغيرات فوق الجينية عبر كامل الجينوم.[13] مصطلح «شيفرة جينية» تم تكييفه كذلك إلى «شيفرة فوق جينية» والتي تستخدم لوصف مجموعة من الميزات فوق الجينية التي تُنشِئ أنماطا ظاهرية مختلفة في خلايا مختلفة تحتوي على نفس تسلسل الدنا. يمكن أن يعبر مصطلح «الشيفرة فوق الجينية» على الحالة الإجمالية للخلية، مع الأخذ في الحسبان موضع كل جزيء في خريطة فوق جينية، تمثيل تخطيطي للتعبير عن الجين، حالة مثيلة الدنا وتعديل الهستون في منطقة جينومية محددة. يُستخدم المصطلح نموذجيا للإشارة إلى مجهودات نظامية لقياس نماذج معلومات فوق جينية محددة ذات صلة مثل شيفرة الهستون وأنماط مثيلة الدنا.
علم النفس النمائي
[عدل]في معنى غير مرتبط نوعا ما بفروع علم الأحياء، يُستخدم مصطلح «علم الجينات الفوقي» في علم النفس النمائي لوصف النمو النفسي كنتيجة للتبادل مزدوج الاتجاه الجاري بين الوراثة والمحيط.[23] نوقشت أفكار تفاعلية حول النمو في نماذج مختلفة وتحت مسميات مختلفة خلال القرنين التاسع عشر والعشرين. اقتُرح نموذج أولي بواسطة كارل إرنست فون باير وشهَّره إرنست هيكل. طُورت نظرة فوق جينية راديكالية (تخلق فوقي فيسيولوجي) بواسطة بول وينتربرت، وعُرض نموذج آخر (تخلق فوقي احتمالي) بواسطة غيلبرت غوتليب في 2003.[24] وتشمل هذه النظرة جميع عوامل النمو الممكنة في كائنٍ وكيف أنها لا تؤثر على الكائن وبعضها البعض فحسب بل تؤثر كذلك على نموه.
كتب عالم النفس النمائي إريك إريكسون حول مبدأٍ فوق جيني في كتابه الهوية: الشباب والأزمة 1968 يشمل فكرة أننا ننمو ونتطور من خلال تكشُّفٍ لشخصيتنا في مراحل محددة مسبقا، وأن بيئتنا وثقافتنا المحيطة بنا تؤثر على كيفية تقدمنا عبر هذه المراحل. يحدث هذا التكشف البيولوجي المرتبط بالخلفيات الاجتماعية-الثقافية في مراحل من النمو النفسي الاجتماعي، أين «يُحدَّد التقدم نحو كل مرحلة بشكل جزئي بواسطة نجاحنا -أو فشلنا- في جميع المراحل السابقة».[25][26][27]
رغم أن الدراسات التجريبية أظهرت نتائج متناقضة، يُعتقد أن التعديلات فوق الجينية هي آلية بيولوجية لصدمة منتقلة بين الأجيال.
الأساس الجزيئي
[عدل]تعدِّل التغيرات فوق الجينية نشاط بعض الجينات، وليس تسلسل الشيفرة الجينية للدنا. يمكن أن تُعدَّل البنية الدقيقة للدنا في حد ذاته أو البروتينات المرتبطة به لتنشيط للجينات أو إسكاتها. تسمح هذه الآلية بتمايز الخلايا في الكائنات متعددة الخلايا للتعبير عن الجينات الضرورية لنشاطها فقط. يُحافظ على التغيرات فوق الجينية حين تنقسم الخلية، وتحدث معظم هذه التغيرات فوق الجينية خلال عمر الكائن الحي، لكن يمكن أن تنتقل التغيرات فوق الجينية إلى نسل الكائن عبر عملية تسمى الوراثة فوق الجينية عبر الأجيال. إذا ظهر تعطيل جين في النطفة أو البويضة التي تُحدث التخصيب، فإن ذلك التغيير فوق الجيني يمكن أن ينتقل كذلك إلى الجيل التالي.[28]
تشمل العمليات فوق الجينية: الطفرات الزائفة، التعليم المرجعي، التطبع الجيني، إسكات الجين، تعطيل الصبغي X، تأثير الموضع، إعادة برمجة مثيلة الدنا، النقل البيني، التأثيرات الأمومية، تقدم التسرطن، التاثيرات العديدة للماسخات، تنظيم تعديلات الهستون والكروماتين المغاير، والحدود التقنية التي تؤثر على التوالد البكري والاستنساخ.
تضرر الدنا
[عدل]يمكن لتضرر الدنا أن يُحدث تغيرات جينية كذلك.[29][30][31] تضرر الدنا شائع جدا، ويحدث بمتوسط حوالي 60 ألف مرة في اليوم لكل خلية من جسم الإنسان (انظر تضرر الدنا الطبيعي) يتم إصلاح هذه الأضرار بالكامل تقريبا، لكن يمكن أن تبقى تغيرات فوق جينية في موقع ترميم الدنا.[32] بالتحديد، يمكن لشرخ في سلسلة الدنا المزدوجة أن يبدأ إسكات جين فوق جيني غير مبرمج عبر التسبب في كل من مثيلة الدنا وتحفيز أنواع الإسكات المتعلقة بتعديل الهستون (تغيير بنية الكروماتين).[33] فضلا على ذلك، يتراكم الإنزيم بوليميراز عديد الأدينوسين ثنائي الفوسفات ريبوز (Parp1) وناتجه عديد الأدينوسين ثنائي الفوسفات ريبوز (PAR) في مواقع تضرر الدنا كجزء من عملية الترميم.[34] يدير هذا التراكم بدوره توظيف وتنشيط بروتين إعادة تغيير بينة الكروماتين ALC1 الذي يمكن أن يُحدث إعادة هيلكة للجسيم النووي.[35] وُجد أن إعادة هيكلة الجسيم النووي تسبب إسكاتا فوق جيني لجين ترميم الدنا MLH1.[22][36] تسبب المواد الكيميائية المضرة للدنا مثل البنزين، هيدروكينون، ستايرين، رباعي كلوريد الكربون وثلاثي كلورو الإيثيلين ضعف مثيلة الدنا، بعضها عبر تفعيل مسارات إجهاد تأكسدي.[37]
معروف بأن الأغذية تغيِّر التأثيرات فوق الجينية للجرذان الذين يتناولون حميات غذائية مختلفة.[38] بعض مكونات الأطعمة تزيد فوق جينيا مستويات إنزيمات ترميم الدنا مثل: O-6-ميثيل غوانين- ناقلة ميثيل الدنا (MGMT) وMLH1[39] وبي53.[40][41] بعض مكونات الأطعمة الأخرى يمكن أن تخفض تضرر الدنا مثل إيزوفلافون الصويا. في أحد الدراسات، انخفضت وسوم الإجهاد التأكسدي -مثل النوكليوتيدات المعدلة التي يمكن أن تسبب تضرر الدنا- بنظام تغذية يحتوي على فول الصويا مدته ثلاث أسابيع.[42] لوحظ كذلك انخفاض تضرر الدنا التأكسدي بعد ساعتين من استهلاك مستخلس ثجير أويسة غني بالأنثوسيان (أويسية آسية).[43]
تقنيات استخدمت لدراسة علم الجينات الفوقي
[عدل]يُستخدم في دراسة علم الجينات الفوقي مجموعة متنوعة من التقنيات الجزيئية الحيوية وذلك لفهمٍ أفضلٍ لظاهرة فوق الجينات، ومنها: الترسيب المناعي للكروماتين (مع التقنيين البديلتين واسعتا النطاق شيب على شيب وشيب-سيك)، التهجين الموضعي المتألق، إنزيمات الاقتطاع الحساسة للمثيلة، تحديد ناقلة ميثيل أدينين الدنا [الإنجليزية] وسلسلة بيكبريتيت [الإنجليزية].[44] علاوة على ذلك، استخدام طرق المعلوماتية الحيوية له دور في علم الجينات الفوقي الحاسوبي.[44]
الآليات
[عدل]هناك عدّة آليات وراثيّة فوق جينيّة، لكن من الجدير بالذكر التنويه إلى أنّ بعضها ليس مقبولًا كليًّا في الأوساط العلميّة.[45]
التعديلات التساهمية للحمض النوويّ والهستون
[عدل]إنّ الكروماتين مكّون من الحمض النوويّ DNA بالإضافة إلى بروتينات الهستون المرتبطة به، فإذا اختلفت الطريقة التي يرتبط بها الحمض مع هذه البروتينات، قد يختلف تمظهر الجينات أيضًا، وتحدث التعديلات على الكروماتين من خلال طريقتين أساسيّتين:
- تعديلات ما بعد الترجمة Posttranslational modifications للأحماض الأمينيّة لبروتينات الهستون، ممّا يؤثّر على ارتباط الحمض النوويّ بها.
- إضافة مجموعات الميثيل الوظيفيّة للحمض النوويّ DNA ممّا يجعل المواقع الممثيلة أقلّ عرضةً لتمظهر جيناتها ويحدث عادةً للسلاسل المكرّرة في الحمض.[46]

الحمض النوويّ الريبوزيّ RNA المنسوخ والبروتينات المشفّرة
[عدل]تنتج بعض الجينات مركبات معينة تساعد على إبقاء نشاطها مستمرًّا، على سبيل المثال فإنّ Hnf4 و MyoD هي مركبات تساعد على تمظهر العديد من الجينات الخاصّة بنشاط الكبد والعضلات، ويعطي الحمض النوويّ الريبوزيّ إشارات تتضمن استدعاءً محددًا لمجموعة من البروتينات لتعديل الكروماتين أيضًا، وإنزيمات ناقلة لمجموعة الميثيل للحمض النوويّ DNA إلى مواقع محدّدة[47] كما تقوم سلاسل مقتطعة من الحمض النوويّ الريبوزي بتغيّرات أخرى فوق جينية عن طريق تشكيل سلاسل مزدوجة من الحمض النوويّ الريبوزيّ RNA. وهذه التغيّرات سيتمّ وراثتها من قبل الجيل التالي حتى لو لم يعد المحفّز الأصلي لتنشيطها موجودًا، هذه الجينات تنشّط وتثبّط عادة عن طريق انتقال الإشارات Signal Transduction، لكنّها قد تنتقل أيضًا عبر الانتشار البسيط من خلال فراغات الفواصل Gap junctions بين الخلايا، وتورّث الأمّ جزءًا كبيرًا من الحمض النوويّ الريبوزيّ والبروتين للبويضة المخصّبة، ممّا ينتج تأثيرًا أموميًّا على الأنماط الظاهريّة، بينما ينتقل جزء صغير من الحمض النوويّ الريبوزيّ من الأبّ إلى البويضة المخصّبة، لكن هناك أدلّة جديدة تظهر بأنّنا نستطيع رؤية تغيّرات واضحة ناتجة من هذا الانتقال على مدى الأجيال المختلفة من الذرّيّة.[48]
الأحماض النوويّة الريبوزيّة الدقيقة MicroRNAs
[عدل]وهي مجموعة من من الأحماض غير المشفِّرة، تتراوح أطوالها بين 17 إلى 25 نيوكليوتيد، وتتحكّم بعمليّات حيويّة كثيرة في النباتات والحيوانات[49] وقد تمّ اكتشاف 2000 نوع منها إلى عام 2013 في الجسم البشريّ وحده،[50] وكل ّواحد من هذه الأحماض قد يستهدف 100-200 حمض ريبوزي رسول mRNA، حيث يكون مسؤولا عن تثبيطه[51] ومعظم هذا التثبيط يحصل من خلال تحلّل الحمض الريبوزيّ الرسول المُستهدَف، لكنّ البعض الآخر يحدث في مرحلة ترجمة الحمض إلى بروتينات.[52] ويبدو لنا أنّ هذه الأحماض الدقيقة (التي يتم التحكّم بها بطريقة فوق جينيّة) تتحكّم بأكثر من 60% من الجينات المشفّرة التي تتمظهر ثمّ تترجم إلى بروتينات[53]، ومن الطرق المقترحة لكيفيّة تثبيط هذه الأحماض فوق جينيًا هي المثيلة لل CpG islands المرتبطة بهذه الأحماض،[54] بالإضافة إلى وجود أحماضٍ أخرى يتمّ تثبيطها عن طريق تعديلات الهستون أو مثيلة الحمض النوويّ DNA المركبة.[49]
الحمض النوويّ الريبوزيّ الرسول
[عدل]في عام 2011، تمّتِ البرهنة على أنّ مثيلة هذا الحمض تلعب دورًا مهمًّا جدًا في التوازن الحراريّ في الجسم البشريّ، كما أنّ الجين المرتبط بالسّمنة (FTO gene) يُحدث تغييرات على مستوى هذا الحمض.[55][56]
الأحماض النوويّة الريبوزيّة الصغيرة RNAs
[عدل]وهي أحماض صغيرة، يترواح طولها بين 50-250 نيكليوتيد، وهي غير مشفّرة وتتواجد في البكتيريا، وظيفتها الرئيسية هي التحكّم بتمظهر الجينات، ويُنظر لها كأداة فاعلة في الصراع مع البكتيريا المقاومة للأدوية المتاحة[57] وتلعب دورًا مهمًّا في الكثير من العمليّات الحيويّة مثل الارتباط بالحمض النوويّ الريبوزيّ الرسول والبروتينات في غير حقيقات النواة (Prokaryotes).
البريونات Prions
[عدل]وهي بروتينات مُعدِيَة، تخرج عن الوظيفة الأساسية للبروتينات التي تقوم بوظائف خلويّة محدّدة، حيث تستطيع تحويل الأشكال الأصليّة للبروتينات إلى شكل آخر مُعْدٍ ومُؤذٍ، وتُعتَبر بهذه الصورة عاملٌ فوق جينيّ يستطيع إحداث تغيير للنمط الظاهري دون تغيير في الجينوم.[58] ويَعتبِر البعض أنّ البريونات الفطريّة عواملًا فوق جينيّة لأنّ النمط الظاهري المعدي منها قد يتمّ وراثته دون تعديل في الجينوم، ويُعتَبر البروتينان (PSI+) و (URE3) اللذان اكتُشِفا في الخميرة عامي 1965 و 1971 أفضل مثالين على هذا النوع من البريونات[59][60] وتعمل هذه البريونات من خلال تحطيم البروتينات المتجمّعة، مقلّلة من نشاطها. وفي الخلايا التي تحتوي على البروتين PSI+ يحدث خلل في البروتين Sup35 مسبّبا معدلًا أعلى من القراءة لدى الرايبوسومات لشيفرة التوقّف.[61][62][63][64][65]
أنظمة الوراثة البنائيّة
[عدل]حيث تُستخدَم البناءات الموجودة للخلايا لصناعة بناءات جديدة في الأجيال القادمة، أمّا الآليات فما زالت بعد غير واضحة للآن.[66][67][68]
تموقع الأجسام النوويّة
[عدل]يتمّ تعليب الجينوم (packaging) بمساعدة الأجسام النوويّة، وتموقع هذه الأجسام ليس عشوائيًا، بل يحدّد إمكانية وصول الحمض النوويّ DNA للبروتينات الناظمة والمتحكّمة، وهذا يحدّد الاختلافات في تمظهر الجينات وتمايز الخلايا، ويتمّ استبقاء بعض الأجسام النوويّة هذه في خلايا الحيوان المنوي. وهكذا، فإنّ تموقع هذه الأجسام قابل للوراثة نسبيًا، وأفادت دراسات حديثة أنّ هناك علاقة بين هذا التموقع وبعض العوامل فوق الجينيّة الأخرى، كمثيلة الحمض النوويّ DNA.[69]
الوظائف والعواقب (Functions and consequences)
[عدل]
النموّ (Development)
[عدل]الوراثة فوق الجينيّة خاصّة عن طريق التعديلات التساهميّة بين الحمض النوويّ DNA والهيستون وتموضع الأجسام النوويّة، هي مهمّة جدًا في تطوّر الكائنات الحيّة متعدّدة الخلايا.[69] إنّ التعديلات فوق الجينية تتحكّم مثلًا بتحوّل الخلايا الجذعية العصبية إلى خلايا دبقية (Glial cells)عن طريق مثيلة الهستون.[70] أمّا النباتات فتتأثر أيضًا بعوامل وعمليّات فوق جينيّة كتعديل الكروماتين، لكنّ بعضها لا يحتاج أو يستخدم «ذاكرة خلوية»، بل تستخدم المعلومات الموقعيّة من البيئة والمحيط من حولها لتحدّد مصيرها.[71]
يُقَسَّم علم ما فوق الجينات علم ما فوق الجينات إلى تخلّق محدّد مسبقًا، وتخلّق محتمل. أمّا الأول فهو طريق ذو اتجاه واحد من التطوّر البنائيّ للحمض النوويّ DNA إلى النضج الوظيفيّ للبروتينات الناتجة، أمّا النوع الثاني (المحتمل)، فهو طريق تطوّر ذو اتجاهين بين البناء والوظيفة.[72]
الطبّ
[عدل]لعلم ما فوق الجينات تطبيقات متنوعة في عالم الطبّ[73] ومن الواضح مثلًا أن لهذا العلم دورًا في فهم الأمراض الجينيّة الخلقيّة، كأمراض Angelman syndrome و Prader-Willi syndrome حيث يتسبّب إلغاء أو تثبيط بعض الجينات في هذين المرضين، لكنّ الغريب هو أنّ هذين المرضين شائعان على غير المتوقع، وما يفسرّ لنا هذا الشيوع هو كون الأفراد المصابين فرادنيّي الزيجوت (Hemizygous) بسبب عمليّة تطبع الجينات (Genomic imprinting)التي هي عمليّة فوق جينيّة بالأساس.
التطوّر (Evolution)
[عدل]من الممكن للعوامل فوق الجينيّة أن تؤثر في التطوّر إذا كانت وراثيّة، واختلف علماء البيولوجيا بين مطالب بتطوير الإطار التصوريّ الأساسيّ للبناء التطوريّ الحديث (modern evolutionary synthesis)[74][75] وبين من ضمّ الوراثة فوق الجينيّة إلى التصوّر الجينيّ السكانيّ (Population genetics)[76] وبين مَنْ ظلّ متشككًا وفاتحًا للاحتمالات على مصراعيها.[77] هناك فرقان أساسيان تختلف بهما الوراثة فوق الجينيّة عن الجينيّة، مع عواقب مهمّة بالنسبة للتطوّر، وهما أنّ معدّل الطفرات فوق الجينيّة أعلى، وأنّ إمكانيّة الرجوع عنها أسهل.[78][79] في النباتات، طفرات مثيلة الحمض النوويّ تحدث بمعدل 100000 مرّة أكثر من الطفرات الجينيّة.[80]
اكتشافات بحثيّة حديثة وأمثلة على التأثيرات
[عدل]تمّ ملاحظة بعض التغيّرات فوق الجينيّة لدى التعرّض لمؤثرات بيئيّة معيّنة. فمثلا، تغيّر لون فرو بعض الفئران وأوزانهم وقابليتهم لنشوء السرطان في أجسادهم لدى تعرّضهم لحميّة غذائيّة معيّنة، من خلال تنشيط جين معيّن Agouti gene.[81][82]
كما أنّ هناك دراسة تقول بأنّ الأحداث الصّادمة قد تولّد مشاعر خوف تُمَرّر للأجيال القادمة عن طريق العوامل فوق الجينيّة مثلاً، أفادت دراسة على الفئران عام 2013 أنّ الفئران قد تنتج ذرّية لديها نفور عامّ من أدوات معيّنة؛ لأنّها كانت مصدرًا لتجارب سيئة لأجدادهم أو آبائهم.[83][84] لكنّ الدراسة السابقة واجهت عدّة انتقادات، منها التحيّز في رصد النتائج بسبب الجودة الإحصائيّة المنخفضة للدراسة[85] كما أنّ عدد الفئران في التجربة قليل لكي يتمّ تعميم النتيجة على البقيّة.[86][87]
أمّا في البشر، فلم يجد الباحثون فرقًا في التوائم المتطابقين (المتعرضين لتأثير بيئي مختلف) في سنين حياتهم الأولى، لكنّ الفرق فوق الجيني اتّضح عندما كبروا قليلًا، حيث ظهر الاختلاف في مثيلة الحمض النوويّ وتعديلات الهستون، وكان التوائم الذين قضوا أقلّ وقت مع بعضهم هم الأكثر اختلافًا على الصعيد فوق الجينيّ.[88] تمّ تسجيل أكثر من 100 ظاهرة وراثة فوق جينيّة عبر الأجيال في طيف واسع من الكائنات الحية (وحيدات الخلايا، النباتات، الحيوانات)[89] على سبيل المثال، تغيّر فراشات Mourning Cloack لونها من خلال تغيّرات معينة في الهرمونات نتيجة تعرّضها لدرجات حرارة مختلفة.[90] كما أظهرت دراسات حديثة إن الإنزيماتنازعات الأمين من عائلة APOBEC/AID من الممكن أن تؤثّر في الوراثة الجينيّة وفوق الجينيّة بشكل متزامن، باستخدام آليات جزيئيّة مشابهة.[91]
تأثيرات فوق جينيّة في البشر
[عدل]تطبّع الجينوم والاضطرابات المتعلقة به (Genomic imprinting and related disorders)
[عدل]وهي ظاهرة في الثدييات حيث يقوم كل من الأبّ والأمّ بتوريث نمط فوق جينيّ مختلف لمواقع معينة في الجينوم في خلاياهم الجنسيّة[92] وأفضل مثالين على ذلك هما: Angelman syndrome و Prader-Willi syndrome. حيث ينتجان بسبب نفس الطفرة الجينية في الكروموسوم الخامس عشر، أمّا الاضطراب الناتج فيعتمد على مصدر الطفرة الموروثة، من الأبّ أو من الأمّ[93] وهذا بسبب وجود تطبّع للجينوم في تلك المنطقة منه. أمّا Beckwith-Wiedmann syndrome فهي ناتجة من تطبّع في الجينوم في الكروموسوم الحادي عشر وهي موروثة من ناحية الأمّ.
مشاهدات فوق جينية عبر الأجيال
[عدل]في دراسة «أوفركاليكس» لاحظ ماركوس بيمبري وزملاؤه أنّ الأحفاد من جهة الأبّ (وليس الذين من جهة الأمّ) للرجال السويديين الّذين تعرّضوا وهم صغار للمجاعة في القرن التاسع عشر، هم أقلّ عُرضة للموت نتيجة مرض في القلب، أمّا لو كان الطعام موجودًا بكثرة، فقد رأوا زيادة ملاحظة لنسب الموت بسبب السكّري لدى الأحفاد، وهذا قد يعطينا دليلًا على تغيّر فوق جينيّ موروث.[94] وعلى عكس ذلك، فقد وجدوا أنّ الحفيدات من جهة الأبّ (وليس اللواتي من جهة الأمّ) للنساء السويديات اللواتي تعرّضن للمجاعة وهنّ ما زالوا في الرّحم، عاشوا حياة أقصر كمعدّل عام.[95]
السرطان والشذوذات التطوّرية
[عدل]ثمّة العديد من المركبات التي تعتبر مسرطنات فوق جينيّة، حيث تقوم هذه المركبات بزيادة معدّل حدوث السرطان دون أن تُحدث طفرة جينيّة مباشرة، ومن هذه المركبات: ثنائي-إيثيل الستيلبيسترول (Diethylstilbestrol)، الأرسينيت (Arsenite)، سداسي كلوروالبنزين (Hexachlorobenzene)، مركبات النيكل (Nickel compunds). الكثير من الماسخات (teratogens) تؤثّر على الأجنّة عبر آليات فوق جينيّة.[96][97] من الممكن أن يظلّ هذا الأثر ويستمرّ طوال حياة الطفل المتأثّر، لكنّ إمكانية أن تنتج تشوّهات خلقيّة نتيجة تعرّض الآباء أو في الجيل الثاني من الذرّية رُفضت نظريًا ونتيجة نقص الأدلّة عليها.[98][99] منظمة الأغذية والأدوية الأمريكية صنّفت دواء (Vidaza) وهو أحد مركبات ال azacitidine، كدواء خطر على الرجال الآباء، وتحذّر من أنّ تناوله يجب ألّا يترافق مع الاقتراب الشديد من الطفل؛ لأنّ تجاربًا على الفئران أثبتت أنّ هذا يقلّل من الخصوبة ويزيد من إمكانيّة إسقاط الجنين، والشذوذات التطوريّة.[100] في الفئران، تمّ رصد اختلافات في نشاط الغدد الصمّ في الذكور المعرّضين للمورفين[101]، وفي الفئران أيضًا، تمّ رصد تغيّرات فوق جينيّة في الجيل الثاني نتيجة التعرّض لثنائي-إيثيل الستيلبيسترول.[102] وتُظهِر دراسات حديثة أنّ ارتباط جين MLL مع جينات أخرى في كروموسومات أخرى يسبّب سرطان الدم (اللوكيميا)، ونعرف أنّ هذا الارتباط يقع تحت سيطرة فوق جينية.[103] كما نتج عن دراسات أخرى أن سرطان البروستات قد يكون مرتبطًا ومتأثرًا بأستلة الهستون (Histone acetylation) ومثيلة الحمض النوويّ DNA، وهي عمليّات فوق جينيّة[104] كما أنّ تمظهر الجينات في البروستات يمكن التحكّم به نسبيًا عن طريق نوعيّة الغذاء المتناول وطريقة الحياة.[105]
مثيلة الحمض النوويّ DNA في السرطان
[عدل]مثيلة الحمض النوويّ هو عامل متحكّم ومهمّ بتمظهر الجينات، كما أنّ هناك أدلّة متزايدة تؤكّد ارتباطه بإخماد الجينات، حيث أنّ الجينات الغنيّة بمركب 5-ميثيل سيتوسين (5-methylcytosine) تكون مُخمَدة، مثيلة الحمض النووي أيضًا عمليّة مهمّة جدًا لتطوّر الجنين، والشذوذات الموجودة في عمليّة المثيلة هذه تمّ ربطها بالعديد من السرطانات، وهي تأتي على شكلين: مثيلة أكثر من اللازم، ومثيلة أقلّ من اللازم، وكلاهما يؤثّر في تطوّر السرطان عبر آليات مختلفة ومتنوعة.[106]
مُصلِحات الحمض النوويّ والسرطان وما فوق الجينات
[عدل]إنّ السرطانات التي تحدث نتيجة طفرات في ال Germ lines في الجينات المصلحة للحمض النوويّ، والتي تصنّف أمراضًا عائلية (familial) تشكّل فقط جزءًا صغيرًا من حالات السرطان.[107] أمّا التغيّرات فوق الجينية التي تحدث تقليلًا في تمظهر الجينات المصلحة للحمض النوويّ، فهي شائعة جدًا في الحالات الفُراديّة من السرطانات بينما تقلّ الطفرات الجينيّة المباشرة في هذه الحالات، [108] كما هو موضّح حسب الجدول التالي:
| المرجع | معدّل الحدوث | التغير فوق الجيني | الجين | السرطان |
| [109] | 13% | CpG island مثيلة | BRCA1 | الصدر |
| [110] | 17% | CpG island مثيلة | WRN | |
| [111] | 36% | CpG island مثيلة | WRN | المبيض |
| [109][112][113] | 5%–30% | CpG island مثيلة | BRCA1 | |
| [112] | 21% | CpG island مثيلة | FANCF | |
| [112] | 3% | CpG island مثيلة | RAD51C | |
| [114][115][116][117][118] | 40%–90% | CpG island مثيلة | MGMT | القولون والمستقيم |
| [110] | 38% | CpG island مثيلة | WRN | |
| [110][115][119] | 2%–65% | CpG island مثيلة | MLH1 | |
| [116] | 13% | CpG islandمثيلة | MSH2 | |
| [120] | 100% | غير معروف | ERCC1 | |
| [120] | 55% | غير معروف | Xpf | |
| [121][122][123][124] | 35%–57% | CpG island مثيلة | MGMT | الرأس والرقبة |
| [125][126][127] | 27%–33% | CpG island مثيلة | MLH1 | |
| [121] | 62% | CpG island مثيلة | NEIL1 | |
| [121] | 46% | CpG island مثيلة | FANCB | |
| [121] | 46% | CpG island مثيلة | MSH4 | |
| [128] | 25% | CpG island مثيلة | ATM |
إنّ المشكلة الرئيسيّة في إحداث خلل في الجينات المصلحة للحمض النوويّ أنّه يسبّب عدم استقرار للجينوم، وهذا هو السبب الرئيسي للتغييرات الجينيّة التي تقود إلى السرطان.[129]
هستون H2A والسرطان
[عدل]
هذا الهستون مهمّ جدًا في الثديات، حيث يلعب أدوارًا عدّة في العمليّات الخلويّة داخل النواة، وأحد أفراد هذه العائلة هو الهستون H2A.X الذي يدلّ على وجود خلل في شريط الحامض النوويّ، وله دور في عملية تصحيح الخلل، ويرتفع هذا الهستون في بعض أنواع السرطانات ويرتبط وجوده بوجود خلل في استقرار جينوم الخلية[130]، وهو مهمّ جدًا في تطوّر الكثير من السرطانات، مثل سرطان الكبد.(156)
علاج السرطان
[عدل]بعض الأبحاث الحديثة تظهر لنا أنّ المستحضرات الدوائيّة فوق الجينيّة قد تكون بديلًا أو علاجًا مضافًا لطرق العلاج التي نستخدمها حاليًا كالإشعاع والعلاج الكيماوي،[131] وإنّ التحكّم بالهستون وأشكاله المختلفة يؤثّر مباشرة على تكون السرطان وتطوّره من عدمه[132] كما إنّ العلاج فوق الجينيّ يمتلك أيضًا خاصيّة مميزة وهي إمكانيّة الرجوع عنه على عكس جميع العلاجات الأخرى.[133] تطوّر البحث في هذه الأدوية استهدف بشكل رئيسيّ الإنزيمات الناقلة للأسيتيل والتي ترتبط بالهستون (Histone acetyltransferase)، وكذلك نازعات الأسيتيل من الهستون (Histone deacetylase)(123)، حيث إنّ الإنزيم الأخير له دور حاسم في تطوّر سرطان الفمّ.[132] ومن الإنزيمات المرشّحة حاليًا لاستهدافها بالعلاج هي ناقلات مجموعة الميثيل المرتبطة بالهستون (Histone lysine methyltranserase) وناقلات الميثيل المرتبطة بالبروتين والأرجينين (protein arginine methyltransferase) .[134][135]
دراسات التوائم
[عدل]أظهرت دراسات حديثة على التوائم المتطابقين وغير المتطابقين أدلّة على تأثير فوق جيني في البشر[88][136] [137] فمِن الممكن أنّ الاختلاف في البيئة يسبّب تأثيرات فوق جينيّة طويلة الأمد، وتطوّرًا مختلفًا.[130] ويبدو أنّ عامل العمر مهمّ جدًا لتراكم الاختلافات فوق الجينيّة بين الأخوين.[88] وأفادت دراسة حديثة على 114 توأم متطابق و80 توأم غير متطابق أنّ انقسامًا متشابهًا للكيسة الأريمية (Blastocyst) بين الأخوين ينتج تشابهًا في الصفات فوق الجينيّة، أي أنّ البيئة الدقيقة المحيطة بالجنين في المرحلة المبكّرة من حياته في الرّحم قد تكون مهمّة جدًا للتأثيرات فوق الجينيّة.[138]
علم ما فوق الجينات في الكائنات الحيّة الدقيقة
[عدل]لدى البكتيريا العديد من العمليّات فوق الجينيّة المشابهة، مثل مثيلة الحمض النوويّ DNA لكن مع بعض الاختلافات في الحمض النوويّ الممثيل (الأدينين بدل السيتوسين) مثيلة الأدينين تعطي إشارةً للتكاثر، وإصلاح الأخطاء، والتحكّم بتمظهر الجينات.[139][140]
ثمّة العديد من المشاريع التي أظهرت قدرتنا على جمع المعلومات فوق الجينيّة من البكتيريا.[141][142][143][144]
في الثقافة الشعبيّة
[عدل]تمّ ذكر علم ما فوق الجينات وتقنيات التحكّم به المستخدمة من قبل البشر للنجاة واستمرار الجنس البشريّ في الحياة، في روايّة للكاتب نيل ستيفنسون، الصادرة عام 2015 واسمها "Seveneves" حيث جاء فيها أنّ البشر ينجون من الأخطار بواسطة عمليّة فوق جينيّة تدعى “going epi”.
ملاحظات
[عدل]- ^ يُعرف المعهد الوطني للصحة والبحث الطبي الفرنسي علم ما فوق الجينات على أنه "فرع من علم الأحياء يدرس طبيعة الآليات التي تغير التعبير الجيني بطريقة قابلة للعكس، انتقالية (أثناء انقسام الخلايا) وتكيفية من دون تغيير تسلسل النوكليوتيدات (الدنا).[5]
« في حين أن علم الجينات يهتم بدراسة الجينات، فإن علم ما فوق الجينات يُعنى بـ "طبقة" من المعلومات التكميلية التي تحدد كيفية استخدام هذه الجينات من قبل خلية أو... عدم استخدامها.[6] »
انظر أيضًا
[عدل]المراجع
[عدل]- ^ Team, Almaany. "ترجمة و معنى epigenetics بالعربي في قاموس المعاني. قاموس عربي انجليزي مصطلحات صفحة 1". www.almaany.com (بالإنجليزية). Archived from the original on 2020-04-11. Retrieved 2020-04-11.
- ^ القاموس الطبي. نسخة محفوظة 19 مايو 2017 على موقع واي باك مشين.
- ^ Dupont C، Armant DR، Brenner CA (سبتمبر 2009). "Epigenetics: definition, mechanisms and clinical perspective". Seminars in Reproductive Medicine. ج. 27 ع. 5: 351–7. DOI:10.1055/s-0029-1237423. PMC:2791696. PMID:19711245.
في المعنى الأصلي لهذا التعريف، يشير علم ما فوق الجينات إلى المسارات الجزيئية التي تعديل التعبير عن نمط جيني إلى نمط ظاهري معين. وخلال الأعوام التالية ومع النمو والتطور السريع في علم الجينات، تم حصر معنى المصطلح بقدر كبير، وأصبح تعريف علم ما فوق الجينات المقبول عموما اليوم هو "دراسة التغيرات في وظيفة الجين التي يتم توريثها بالانقسام المتساوي أو المنصف والتي لا تنطوي على تغيُّرٍ في تسلسل الدنا".
- ^ Rutherford، Adam (19 يوليو 2015). "Beware the pseudo gene genies". الغارديان. مؤرشف من الأصل في 2020-03-25.
- ^ "Epigénétique | Inserm". www.inserm.fr (بالفرنسية). Archived from the original on 2021-07-15. Retrieved 2017-11-29.
- ^ Dossier "Epigénétique". le site de l'المعهد الوطني للصحة والبحوث الطبية . فبراير 2015. مؤرشف من الأصل في 2023-02-22. اطلع عليه بتاريخ 2015-09-03.
{{استشهاد ويب}}: صيانة الاستشهاد: علامات ترقيم زائدة (link) نسخة محفوظة 2020-05-24 على موقع واي باك مشين. - ^ ا ب Ledford H (أكتوبر 2008). "Language: Disputed definitions". Nature. ج. 455 ع. 7216: 1023–8. DOI:10.1038/4551023a. PMID:18948925.
- ^ ا ب ج د Berger SL، Kouzarides T، Shiekhattar R، Shilatifard A (أبريل 2009). "An operational definition of epigenetics". Genes & Development. ج. 23 ع. 7: 781–3. DOI:10.1101/gad.1787609. PMC:3959995. PMID:19339683.
- ^ ا ب Bird A (مايو 2007). "Perceptions of epigenetics". Nature. ج. 447 ع. 7143: 396–8. Bibcode:2007Natur.447..396B. DOI:10.1038/nature05913. PMID:17522671.
- ^ Hunter، Philip (1 مايو 2008). "What genes remember". Prospect Magazine. مؤرشف من الأصل في 2008-05-01. اطلع عليه بتاريخ 2012-07-26.
{{استشهاد ويب}}: الوسيط غير المعروف|name-list-format=تم تجاهله يقترح استخدام|name-list-style=(مساعدة) - ^ Reik W (مايو 2007). "Stability and flexibility of epigenetic gene regulation in mammalian development". Nature. ج. 447 ع. 7143: 425–32. Bibcode:2007Natur.447..425R. DOI:10.1038/nature05918. PMID:17522676.
- ^ Moore، David S. (2015). The Developing Genome: An Introduction to Behavioral Epigenetics (ط. 1st). Oxford University Press. ISBN:978-0199922345. مؤرشف من الأصل في 2020-04-11.
- ^ ا ب ج "Overview". NIH Roadmap Epigenomics Project. مؤرشف من الأصل في 2019-11-21.
- ^ [[قاموس أكسفورد الإنجليزي|]]: "The word is used by W. Harvey, Exercitationes 1651, p. 148, and in the English Anatomical Exercitations 1653, p. 272. It is explained to mean ‘partium super-exorientium additamentum’, ‘the additament of parts budding one out of another’."
- ^ Waddington CH (1942). "The epigenotype". Endeavour. ج. 1: 18–20. "لغرض دراسة الوراثة، العلاقة بين الأنماط الظاهرية والأنماط الجينية [...] هي -من وجهة نظر بيولوجية أوسع- ذات أهمية حاسمة لأنها نواة مشكل النمو بالكامل. أدرك العديد من علماء الجينات هذا الأمر وحاولوا اكتشاف العمليات التي لها دور في الآلية التي تُحدث بها جيناتُ نمط جينيٍ تأثيرات النمط الظاهري. الخطوة الأولى في مشروع كهذا هي -أو يجب أن تكون، لأنها غالبا ما تُهمَل من قبل أولائك الذين لديهم احترام مفرط لقوى المنطق- وصف ما يمكن رؤيته من العمليات النمائية. لدراساتٍ من هذا النوع تم صياغة مصطلح علم الجينات الظاهري (phenogenetics) بواسطة هيكر سنة 1918. الجزء الثاني والأهم من المهمة هو اكتشاف الآليات المسببة أثناء عملها، وربطها بأقصى قدر ممكن بما اكتشفه علم الأجنة التجريبي حتى الآن من آليات النمو. ربما نستخدم مصطلح علم مافوق الجينات (epigenetics) لمثل هذه الدراسات -وبالتالي التأكيد على علاقتها مع المفاهيم- المفضِّلة بشدة للنظرية الكلاسيكية للتخلق الفوقي، والتي تم تحقيقها بواسطة الأخصاء في علم الأجنة التجريبي. علينا بالتأكيد التذكر بأن بين النمط الجيني والنمط الظاهري وبين الربط بينهما تتواجد عمليات نمو معقدة، ومن المناسب إطلاق إسم على هذا المعقد، وتسمية 'نمط جيني فوقي' (epigenotype) تبدو مناسبة."
- ^ انظر نظرية التكون المسبق من أجل خلفية تاريخية. قاموس أكسفورد الإنجليزي: نظرة أن الجنين يظهر إلى الوجود (تنامٍ تراكمي متوالي أو متعاقب) وليس مجرد نمو، في عملية التكاثر. [...] النظرية المضادة كانت تُعرف سابقة 'بنظرية التطور' ولتجنب الالتباس أصبح يُشار إليها أساسا 'بنظرية التكون المسبق' وأحيانا 'بنظرية التغليف' (encasement) أو الإدغام والتجميع (emboîtement).
- ^ Waddington، C. H. (2014). The Epigenetics of Birds. Cambridge University Press. ISBN:978-1-107-44047-0. مؤرشف من الأصل في 2020-06-03.[بحاجة لرقم الصفحة]
- ^ Hall BK (يناير 2004). "In search of evolutionary developmental mechanisms: the 30-year gap between 1944 and 1974". Journal of Experimental Zoology Part B: Molecular and Developmental Evolution. ج. 302 ع. 1: 5–18. DOI:10.1002/jez.b.20002. PMID:14760651.
- ^ Alvarez-Buylla ER، Chaos A، Aldana M، Benítez M، Cortes-Poza Y، Espinosa-Soto C، وآخرون (3 نوفمبر 2008). "Floral morphogenesis: stochastic explorations of a gene network epigenetic landscape". PLOS ONE. ج. 3 ع. 11: e3626. Bibcode:2008PLoSO...3.3626A. DOI:10.1371/journal.pone.0003626. PMC:2572848. PMID:18978941.
- ^ ا ب Rabajante JF، Babierra AL (مارس 2015). "Branching and oscillations in the epigenetic landscape of cell-fate determination". Progress in Biophysics and Molecular Biology. ج. 117 ع. 2–3: 240–249. DOI:10.1016/j.pbiomolbio.2015.01.006. PMID:25641423.
- ^ Holliday R (يناير 1990). "DNA methylation and epigenetic inheritance". Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences. ج. 326 ع. 1235: 329–38. Bibcode:1990RSPTB.326..329H. DOI:10.1098/rstb.1990.0015. PMID:1968668.
- ^ ا ب Riggs AD، Martienssen RA، Russo VE (1996). Epigenetic mechanisms of gene regulation. Plainview, NY: Cold Spring Harbor Laboratory Press. ص. 1–4. ISBN:978-0-87969-490-6.[بحاجة لرقم الصفحة]
- ^ Gottlieb G (1991). "Epigenetic systems view of human development". Developmental Psychology. ج. 27 ع. 1: 33–34. DOI:10.1037/0012-1649.27.1.33.
- ^ Gilbert Gottlieb. Probabilistic epigenesis, Developmental Science 10:1 (2007), 1–11 نسخة محفوظة 2017-08-28 على موقع واي باك مشين.
- ^ Boeree, C. George, (1997/2006), Personality Theories, Erik Erikson نسخة محفوظة 2020-03-27 على موقع واي باك مشين.
- ^ Erikson، Erik (1968). Identity: Youth and Crisis. Chapter 3: W.W. Norton and Company. ص. 92. مؤرشف من الأصل في 2020-06-03.
{{استشهاد بكتاب}}: صيانة الاستشهاد: مكان (link) - ^ "Epigenetics". Bio-Medicine.org. مؤرشف من الأصل في 2019-11-18. اطلع عليه بتاريخ 2011-05-21.
- ^ Chandler VL (فبراير 2007). "Paramutation: from maize to mice". Cell. ج. 128 ع. 4: 641–5. DOI:10.1016/j.cell.2007.02.007. PMID:17320501.
- ^ Kovalchuk O، Baulch JE (يناير 2008). "Epigenetic changes and nontargeted radiation effects--is there a link?". Environmental and Molecular Mutagenesis. ج. 49 ع. 1: 16–25. DOI:10.1002/em.20361. PMID:18172877.
- ^ Ilnytskyy Y، Kovalchuk O (سبتمبر 2011). "Non-targeted radiation effects-an epigenetic connection". Mutation Research. ج. 714 ع. 1–2: 113–25. DOI:10.1016/j.mrfmmm.2011.06.014. PMID:21784089.
- ^ Friedl AA، Mazurek B، Seiler DM (2012). "Radiation-induced alterations in histone modification patterns and their potential impact on short-term radiation effects". Frontiers in Oncology. ج. 2: 117. DOI:10.3389/fonc.2012.00117. PMC:3445916. PMID:23050241.
{{استشهاد بدورية محكمة}}: صيانة الاستشهاد: دوي مجاني غير معلم (link) - ^ Cuozzo C، Porcellini A، Angrisano T، Morano A، Lee B، Di Pardo A، وآخرون (يوليو 2007). "DNA damage, homology-directed repair, and DNA methylation". PLOS Genetics. ج. 3 ع. 7: e110. DOI:10.1371/journal.pgen.0030110. PMC:1913100. PMID:17616978.
{{استشهاد بدورية محكمة}}: صيانة الاستشهاد: دوي مجاني غير معلم (link) - ^ O'Hagan HM، Mohammad HP، Baylin SB (أغسطس 2008). Lee JT (المحرر). "Double strand breaks can initiate gene silencing and SIRT1-dependent onset of DNA methylation in an exogenous promoter CpG island". PLOS Genetics. ج. 4 ع. 8: e1000155. DOI:10.1371/journal.pgen.1000155. PMC:2491723. PMID:18704159.
{{استشهاد بدورية محكمة}}: صيانة الاستشهاد: دوي مجاني غير معلم (link) - ^ Malanga M، Althaus FR (June 2005). "The role of poly(ADP-ribose) in the DNA damage signaling network" (PDF). Biochemistry and Cell Biology. ج. 83 ع. 3: 354–64. DOI:10.1139/o05-038. PMID:15959561. مؤرشف من الأصل (PDF) في 4 يونيو 2020. اطلع عليه بتاريخ أكتوبر 2020.
{{استشهاد بدورية محكمة}}: تحقق من التاريخ في:|تاريخ الوصول=(مساعدة) - ^ Gottschalk AJ، Timinszky G، Kong SE، Jin J، Cai Y، Swanson SK، وآخرون (أغسطس 2009). "Poly(ADP-ribosyl)ation directs recruitment and activation of an ATP-dependent chromatin remodeler". Proceedings of the National Academy of Sciences of the United States of America. ج. 106 ع. 33: 13770–4. Bibcode:2009PNAS..10613770G. DOI:10.1073/pnas.0906920106. PMC:2722505. PMID:19666485.
- ^ Lin JC، Jeong S، Liang G، Takai D، Fatemi M، Tsai YC، وآخرون (نوفمبر 2007). "Role of nucleosomal occupancy in the epigenetic silencing of the MLH1 CpG island". Cancer Cell. ج. 12 ع. 5: 432–44. DOI:10.1016/j.ccr.2007.10.014. PMC:4657456. PMID:17996647.
- ^ Tabish AM، Poels K، Hoet P، Godderis L (2012). Chiariotti L (المحرر). "Epigenetic factors in cancer risk: effect of chemical carcinogens on global DNA methylation pattern in human TK6 cells". PLOS ONE. ج. 7 ع. 4: e34674. Bibcode:2012PLoSO...734674T. DOI:10.1371/journal.pone.0034674. PMC:3324488. PMID:22509344.
{{استشهاد بدورية محكمة}}: صيانة الاستشهاد: دوي مجاني غير معلم (link) - ^ Burdge GC، Hoile SP، Uller T، Thomas NA، Gluckman PD، Hanson MA، Lillycrop KA (2011). Imhof A (المحرر). "Progressive, transgenerational changes in offspring phenotype and epigenotype following nutritional transition". PLOS ONE. ج. 6 ع. 11: e28282. Bibcode:2011PLoSO...628282B. DOI:10.1371/journal.pone.0028282. PMC:3227644. PMID:22140567.
{{استشهاد بدورية محكمة}}: صيانة الاستشهاد: دوي مجاني غير معلم (link) - ^ Fang M، Chen D، Yang CS (يناير 2007). "Dietary polyphenols may affect DNA methylation". The Journal of Nutrition. ج. 137 ع. 1 Suppl: 223S–228S. DOI:10.1093/jn/137.1.223S. PMID:17182830.
- ^ Olaharski AJ، Rine J، Marshall BL، Babiarz J، Zhang L، Verdin E، Smith MT (ديسمبر 2005). "The flavoring agent dihydrocoumarin reverses epigenetic silencing and inhibits sirtuin deacetylases". PLOS Genetics. ج. 1 ع. 6: e77. DOI:10.1371/journal.pgen.0010077. PMC:1315280. PMID:16362078.
{{استشهاد بدورية محكمة}}: صيانة الاستشهاد: دوي مجاني غير معلم (link) - ^ Kikuno N، Shiina H، Urakami S، Kawamoto K، Hirata H، Tanaka Y، وآخرون (أغسطس 2008). "Genistein mediated histone acetylation and demethylation activates tumor suppressor genes in prostate cancer cells". International Journal of Cancer. ج. 123 ع. 3: 552–60. DOI:10.1002/ijc.23590. PMID:18431742.
- ^ Djuric Z، Chen G، Doerge DR، Heilbrun LK، Kucuk O (أكتوبر 2001). "Effect of soy isoflavone supplementation on markers of oxidative stress in men and women". Cancer Letters. ج. 172 ع. 1: 1–6. DOI:10.1016/S0304-3835(01)00627-9. PMID:11595123.
- ^ Kropat C، Mueller D، Boettler U، Zimmermann K، Heiss EH، Dirsch VM، وآخرون (مارس 2013). "Modulation of Nrf2-dependent gene transcription by bilberry anthocyanins in vivo". Molecular Nutrition & Food Research. ج. 57 ع. 3: 545–50. DOI:10.1002/mnfr.201200504. PMID:23349102.
- ^ ا ب Verma M، Rogers S، Divi RL، Schully SD، Nelson S، Joseph Su L، وآخرون (فبراير 2014). "Epigenetic research in cancer epidemiology: trends, opportunities, and challenges". Cancer Epidemiology, Biomarkers & Prevention. ج. 23 ع. 2: 223–33. DOI:10.1158/1055-9965.EPI-13-0573. PMC:3925982. PMID:24326628.
- ^ Jablonka E, Lamb MJ, Lachmann M (September 1992). "Evidence, mechanisms and models for the inheritance of acquired characteristics". J. Theor. Biol. 158 (2): 245–268.doi:10.1016/S0022-5193(05)80722-2.
- ^ Slotkin RK, Martienssen R (April 2007). "Transposable elements and the epigenetic regulation of the genome". Nature Reviews Genetics 8 (4): 272–85.doi:10.1038/nrg2072. ببمد: 17363976.
- ^ Mattick JS, Amaral PP, Dinger ME, Mercer TR, Mehler MF (January 2009). "RNA regulation of epigenetic processes". BioEssays 31 (1): 51–9. doi:10.1002/bies.080099.ببمد: 19154003.
- ^ Choi CQ (25 May 2006). "The Scientist: RNA can be hereditary molecule". The Scientist. Retrieved 2006.
- ^ ا ب to:a b c Bernal JE, Duran C, Papiha SS (2012). "Transcriptional and epigenetic regulation of human microRNAs". Cancer Lett 331 (1): 1–10. doi:10.1016/j.canlet.2012.12.006.ببمد: 3246373.
- ^ Browse miRBase by species
- ^ Lim LP, Lau NC, Garrett-Engele P, Grimson A, Schelter JM, Castle J et al. (2005). "Microarray analysis shows that some microRNAs downregulate large numbers of target mRNAs". Nature 433 (7027): 769–773. doi:10.1038/nature03315. ببمد: 15685193.
- ^ Lee D, Shin C (2012). MicroRNA-target interactions: new insights from genome-wide approaches" Ann N Y Acad Sci 1271:118-28. doi: 10.1111/j.1749-6632.2012.06745.x. Review. ببمد: 23050973
- ^ Friedman RC, Farh KK, Burge CB, Bartel DP (2009). "Most mammalian mRNAs are conserved targets of microRNAs". Genome Res 19 (1): 92–105.doi:10.1101/gr.082701.108. PMC 2612969. ببمد: 18955434.
- ^ Goll MG, Bestor TH (2005). "Eukaryotic cytosine methyltransferases". Annu Rev Biochem74: 481–514. doi:10.1146/annurev.biochem.74.010904.153721. ببمد: 15952895.
- ^ Guifang Jia; Ye Fu; Xu Zhao; Qing Dai; Guanqun Zheng; Ying Yang; Chengqi Yi; Lindahl, Tomas; Tao Pan; Yun-Gui Yang; Chuan He (16 October 2011). "N6-Methyladenosine in nuclear RNA is a major substrate of the obesity-associated FTO".Nature Chemical Biology 7 (12): 885–887. doi:10.1038/nchembio.687. PMC 3218240.ببمد: 22002720.
- ^ "New research links common RNA modification to obesity". Physorg.com. Retrieved26 July 2012.
- ^ Howden BP, Beaume M, Harrison PF, Hernandez D, Schrenzel J, Seemann T et al. (August 2013). "Analysis of the Small RNA Transcriptional Response in Multidrug-Resistant Staphylococcus aureus after Antimicrobial Exposure". Antimicrob. Agents Chemother. 57(8): 3864–74. doi:10.1128/AAC.00263-13. PMC 3719707. ببمد: 23733475.
- ^ Yool A, Edmunds WJ (1998). "Epigenetic inheritance and prions". Journal of Evolutionary Biology 11 (2): 241–242. doi:10.1007/s000360050085.
- ^ Cox BS (1965). "[PSI], a cytoplasmic suppressor of super-suppression in yeast". Heredity20 (4): 505–521. doi:10.1038/hdy.1965.65.
- ^ Lacroute F (May 1971). "Non-Mendelian mutation allowing ureidosuccinic acid uptake in yeast". J. Bacteriol. 106 (2): 519–22. PMC 285125. ببمد: 5573734.
- ^ Liebman SW, Sherman F (September 1979). "Extrachromosomal psi+ determinant suppresses nonsense mutations in yeast". J. Bacteriol. 139 (3): 1068–71.PMC 218059. ببمد: 225301.
- ^ True HL, Lindquist SL (September 2000). "A yeast prion provides a mechanism for genetic variation and phenotypic diversity". Nature 407 (6803): 477–83.doi:10.1038/35035005. ببمد: 11028992.
- ^ Shorter J, Lindquist S (June 2005). "Prions as adaptive conduits of memory and inheritance". Nature Reviews Genetics 6 (6): 435–50. doi:10.1038/nrg1616.ببمد: 15931169.
- ^ Giacomelli MG, Hancock AS, Masel J (2007). "The conversion of 3′ UTRs into coding regions". Molecular Biology & Evolution 24 (2): 457–464. doi:10.1093/molbev/msl172.PMC 1808353. ببمد: 17099057.
- ^ Lancaster AK, Bardill JP, True HL, Masel J (2010). "The Spontaneous Appearance Rate of the Yeast Prion PSI+ and Its Implications for the Evolution of the Evolvability Properties of the PSI+ System". Genetics 184 (2): 393–400. doi:10.1534/genetics.109.110213.PMC 2828720. ببمد: 19917766.
- ^ Sapp J (1991). "Concepts of organization. The leverage of ciliate protozoa". Dev. Biol. (NY) 7: 229–58. doi:10.1007/978-1-4615-6823-0_11. ببمد: 1804215.
- ^ Sapp J (2003). Genesis: the evolution of biology. Oxford [Oxfordshire]: Oxford University Press. ISBN 0-19-515619-6.
- ^ Gray RD, Oyama S, Griffiths PE (2003). Cycles of Contingency: Developmental Systems and Evolution (Life and Mind: Philosophical Issues in Biology and Psychology). Cambridge, Mass: The MIT Press. ISBN 0-262-65063-0.
- ^ ا ب to:a b Teif VB, Beshnova DA, Vainshtein Y, Marth C, Mallm JP, Höfer T et al. (8 May 2014). "Nucleosome repositioning links DNA (de)methylation and differential CTCF binding during stem cell development". Genome Research 24: 1285–1295. doi:10.1101/gr.164418.113.
- ^ Chapter: "Nervous System Development" in "Epigenetics," by Benedikt Hallgrimsson and Brian Hall
- ^ Costa S, Shaw P (March 2007). "'Open minded' cells: how cells can change fate"(PDF). Trends Cell Biol. 17 (3): 101–6. doi:10.1016/j.tcb.2006.12.005.ببمد: 17194589. This might suggest that plant cells do not use or require a cellular memory mechanism and just respond to positional information. However, it has been shown that plants do use cellular memory mechanisms mediated by PcG proteins in several processes, ... (p.104)
- ^ Griesemer J, Haber MH, Yamashita G, Gannett L (March 2005). "Critical Notice: Cycles of Contingency – Developmental Systems and Evolution". Biology & Philosophy 20 (2–3): 517–544. doi:10.1007/s10539-004-0836-4.
- ^ Chahwan R, Wontakal SN, Roa S (March 2011). "The multidimensional nature of epigenetic information and its role in disease". Discov Med 11 (58): 233–43.ببمد: 21447282.
- ^ Lamb MJ, Jablonka E (2005). Evolution in four dimensions: genetic, epigenetic, behavioral, and symbolic variation in the history of life. Cambridge, Mass: MIT Press.ISBN 0-262-10107-6.
- ^ See also Denis Noble The Music of Life see esp pp. 93–8 and p. 48 where he cites Jablonka & Lamb and Massimo Pigliucci's review of Jablonka and Lamb in Nature 435, 565–566 (2 June 2005)
- ^ Maynard Smith J (1990). "Models of a Dual Inheritance System". Journal of Theoretical Biology 143 (1): 41–53. doi:10.1016/S0022-5193(05)80287-5. ببمد: 2359317.
- ^ Lynch M (2007). "The frailty of adaptive hypotheses for the origins of organismal complexity". PNAS 104 (suppl. 1): 8597–8604. Bibcode:2007PNAS..104.8597L.doi:10.1073/pnas.0702207104. PMC 1876435. ببمد: 17494740.
- ^ Rando OJ, Verstrepen KJ (February 2007). "Timescales of genetic and epigenetic inheritance". Cell 128 (4): 655–68. doi:10.1016/j.cell.2007.01.023. ببمد: 17320504.
- ^ Lancaster AK, Masel J (1 September 2009). "The evolution of reversible switches in the presence of irreversible mimics". Evolution 63 (9): 2350–2362. doi:10.1111/j.1558-5646.2009.00729.x. PMC 2770902. ببمد: 19486147.
- ^ Graaf, Adriaan van der; Wardenaar, René; Neumann, Drexel A.; Taudt, Aaron; Shaw, Ruth G.; Jansen, Ritsert C.; Schmitz, Robert J.; Colomé-Tatché, Maria; Johannes, Frank (2015-05-11). "Rate, spectrum, and evolutionary dynamics of spontaneous epimutations".Proceedings of the National Academy of Sciences 112: 201424254.doi:10.1073/pnas.1424254112. ISSN 0027-8424. Retrieved 2015-05-12.
- ^ Cooney CA, Dave AA, Wolff GL (August 2002). "Maternal methyl supplements in mice affect epigenetic variation and DNA methylation of offspring". J. Nutr. 132 (8 Suppl): 2393S–2400S. ببمد: 12163699.
- ^ Waterland RA, Jirtle RL (August 2003). "Transposable elements: targets for early nutritional effects on epigenetic gene regulation". Mol. Cell. Biol. 23 (15): 5293–300.doi:10.1128/MCB.23.15.5293-5300.2003. PMC 165709. ببمد: 12861015.
- ^ Fearful Memories Passed Down to Mouse Descendants: Genetic imprint from traumatic experiences carries through at least two generations, By Ewen Callaway and Nature magazine | Sunday, 1 December 2013.
- ^ Mice can 'warn' sons, grandsons of dangers via sperm, by Mariette Le Roux, 12/1/13.
- ^ G. Francis, "Too Much Success for Recent Groundbreaking Epigenetic Experiments"http://www.genetics.org/content/198/2/449.abstract نسخة محفوظة 2020-02-28 على موقع واي باك مشين.
- ^ http://www.ncbi.nlm.nih.gov/pubmed/24292232 (see comment by Gonzalo Otazu) نسخة محفوظة 2020-04-04 على موقع واي باك مشين.
- ^ Epigenetics Paper Raises Questions | The Scientist Magazine® نسخة محفوظة 25 مارس 2017 على موقع واي باك مشين.
- ^ ا ب ج to:a b c Fraga MF, Ballestar E, Paz MF, Ropero S, Setien F, Ballestar ML et al. (July 2005)."Epigenetic differences arise during the lifetime of monozygotic twins". Proc. Natl. Acad. Sci. U.S.A. 102 (30): 10604–9. Bibcode:2005PNAS..10210604F.doi:10.1073/pnas.0500398102. PMC 1174919. ببمد: 16009939.
- ^ Jablonka E, Raz G (June 2009). "Transgenerational epigenetic inheritance: prevalence, mechanisms, and implications for the study of heredity and evolution". Q Rev Biol 84 (2): 131–76. doi:10.1086/598822. ببمد: 19606595.
- ^ Davies, Hazel (2008). Do Butterflies Bite?: Fascinating Answers to Questions about Butterflies and Moths (Animals Q&A). Rutgers University Press.
- ^ Chahwan R, Wontakal SN, Roa S (October 2010). "Crosstalk between genetic and epigenetic information through cytosine deamination". Trends Genet. 26 (10): 443–8.doi:10.1016/j.tig.2010.07.005. ببمد: 20800313.
- ^ Wood AJ, Oakey RJ (November 2006). "Genomic imprinting in mammals: emerging themes and established theories". PLoS Genet. 2 (11): e147.doi:10.1371/journal.pgen.0020147. PMC 1657038. ببمد: 17121465.
- ^ A person's paternal grandson is the son of a son of that person; a maternal grandson is the son of a daughter.
- ^ Pembrey ME, Bygren LO, Kaati G, Edvinsson S, Northstone K, Sjöström M et al. (February 2006). "Sex-specific, male-line transgenerational responses in humans". Eur. J. Hum. Genet. 14 (2): 159–66. doi:10.1038/sj.ejhg.5201538. ببمد: 16391557. Robert Winston refers to this study in a lecture; see also discussion at Leeds University, here[1]
- ^ "NOVA | Transcripts | Ghost in Your Genes". PBS. 16 October 2007. Retrieved 26 July2012.
- ^ Bishop JB, Witt KL, Sloane RA (December 1997). "Genetic toxicities of human teratogens". Mutat. Res. 396 (1–2): 9–43. doi:10.1016/S0027-5107(97)00173-5.ببمد: 9434858.
- ^ Gurvich N, Berman MG, Wittner BS, Gentleman RC, Klein PS, Green JB (July 2005). "Association of valproate-induced teratogenesis with histone deacetylase inhibition in vivo".FASEB J. 19 (9): 1166–8. doi:10.1096/fj.04-3425fje. ببمد: 15901671.
- ^ Smithells D (November 1998). "Does thalidomide cause second generation birth defects?". Drug Saf 19 (5): 339–41. doi:10.2165/00002018-199819050-00001.ببمد: 9825947.
- ^ Friedler G (December 1996). "Paternal exposures: impact on reproductive and developmental outcome. An overview". Pharmacol. Biochem. Behav. 55 (4): 691–700.doi:10.1016/S0091-3057(96)00286-9. ببمد: 8981601.
- ^ WebCite query result
- ^ Cicero TJ, Adams ML, Giordano A, Miller BT, O'Connor L, Nock B (March 1991). "Influence of morphine exposure during adolescence on the sexual maturation of male rats and the development of their offspring". J. Pharmacol. Exp. Ther. 256 (3): 1086–93.ببمد: 2005573.
- ^ Newbold RR, Padilla-Banks E, Jefferson WN (June 2006). "Adverse effects of the model environmental estrogen diethylstilbestrol are transmitted to subsequent generations".Endocrinology 147 (6 Suppl): S11–7. doi:10.1210/en.2005-1164. ببمد: 16690809.
- ^ Mandal SS (April 2010). "Mixed lineage leukemia: versatile player in epigenetics and human disease". FEBS J. 277 (8): 1789. doi:10.1111/j.1742-4658.2010.07605.x.ببمد: 20236314.
- ^ to:a b Li LC, Carroll PR, Dahiya R (January 2005). "Epigenetic changes in prostate cancer: implication for diagnosis and treatment". J. Natl. Cancer Inst. 97 (2): 103–15.doi:10.1093/jnci/dji010. ببمد: 15657340.
- ^ Ornish D, Magbanua MJ, Weidner G, Weinberg V, Kemp C, Green C et al. (June 2008)."Changes in prostate gene expression in men undergoing an intensive nutrition and lifestyle intervention". Proc. Natl. Acad. Sci. U.S.A. 105 (24): 8369–74.Bibcode:2008PNAS..105.8369O. doi:10.1073/pnas.0803080105. PMC 2430265.ببمد: 18559852.
- ^ to:a b Wong NC, Craig JM (2011). Epigenetics: A Reference Manual. Norfolk, England: Caister Academic Press. ISBN 1-904455-88-3.
- ^ Jasperson KW, Tuohy TM, Neklason DW, Burt RW (2010). "Hereditary and familial colon cancer". Gastroenterology 138 (6): 2044–2058. doi:10.1053/j.gastro.2010.01.054.ببمد: 20420945.
- ^ Wood LD, Parsons DW, Jones S, Lin J, Sjöblom T, Leary RJ et al. (2007). "The genomic landscapes of human breast and colorectal cancers". Science 318 (5853): 1108–1113.doi:10.1126/science.1145720. ببمد: 17932254.
- ^ ا ب to:a b Esteller M, Silva JM, Dominguez G, Bonilla F, Matias-Guiu X, Lerma E et al. (Apr 2000). "Promoter hypermethylation and BRCA1 inactivation in sporadic breast and ovarian tumors.". J Natl Cancer Inst 92 (7): 564–9. doi:10.1093/jnci/92.7.564.ببمد: 10749912.
- ^ ا ب ج to:a b c Agrelo R, Cheng WH, Setien F, Ropero S, Espada J, Fraga MF et al. (Jun 2006). "Epigenetic inactivation of the premature aging Werner syndrome gene in human cancer.".Proc Natl Acad Sci U S A 103 (23): 8822–7. doi:10.1073/pnas.0600645103.ببمد: 16723399.
- ^ Baldwin RL, Nemeth E, Tran H, Shvartsman H, Cass I, Narod S et al. (2000). "BRCA1 promoter region hypermethylation in ovarian carcinoma: a population-based study". Cancer Res 60 (19): 5329–5333. ببمد: 11034065.
- ^ ا ب ج to:a b c Rigakos G, Razis E (2012). "BRCAness: finding the Achilles heel in ovarian cancer". Oncologist 17 (7): 956–62. doi:10.1634/theoncologist.2012-0028.PMC 3399652. ببمد: 22673632.
- ^ Stefansson OA, Villanueva A, Vidal A, Martí L, Esteller M (2012). "BRCA1 epigenetic inactivation predicts sensitivity to platinum-based chemotherapy in breast and ovarian cancer". Epigenetics 7 (11): 1225–1229. doi:10.4161/epi.22561. ببمد: 23069641.
- ^ Shen L, Kondo Y, Rosner GL, Xiao L, Hernandez NS, Vilaythong J et al. (2005). "MGMT promoter methylation and field defect in sporadic colorectal cancer". J Natl Cancer Inst 97(18): 1330–1338. doi:10.1093/jnci/dji275. ببمد: 16174854.
- ^ ا ب to:a b Psofaki V, Kalogera C, Tzambouras N, Stephanou D, Tsianos E, Seferiadis K et al. (2010). "Promoter methylation status of hMLH1, MGMT, and CDKN2A/p16 in colorectal adenomas". World J Gastroenterol 16 (28): 3553–3560. doi:10.3748/wjg.v16.i28.3553.ببمد: 20653064.
- ^ ا ب to:a b Lee KH, Lee JS, Nam JH, Choi C, Lee MC, Park CS et al. (2011). "Promoter methylation status of hMLH1, hMSH2, and MGMT genes in colorectal cancer associated with adenoma-carcinoma sequence". Langenbecks Arch Surg 396 (7): 1017–1026.doi:10.1007/s00423-011-0812-9. ببمد: 21706233.
- ^ Amatu A, Sartore-Bianchi A, Moutinho C, Belotti A, Bencardino K, Chirico G, Cassingena A, Rusconi F, Esposito A, Nichelatti M, Esteller M, Siena S (2013). "Promoter CpG island hypermethylation of the DNA repair enzyme MGMT predicts clinical response to dacarbazine in a phase II study for metastatic colorectal cancer". Clin. Cancer Res. 19 (8): 2265–72. doi:10.1158/1078-0432.CCR-12-3518. ببمد: 23422094.
- ^ Mokarram P, Zamani M, Kavousipour S, Naghibalhossaini F, Irajie C, Moradi Sarabi M, Hosseini SV (2013). "Different patterns of DNA methylation of the two distinct O6-methylguanine-DNA methyltransferase (O6-MGMT) promoter regions in colorectal cancer".Mol. Biol. Rep. 40 (5): 3851–7. doi:10.1007/s11033-012-2465-3. ببمد: 23271133.
- ^ Truninger K, Menigatti M, Luz J, Russell A, Haider R, Gebbers JO et al. (2005). "Immunohistochemical analysis reveals high frequency of PMS2 defects in colorectal cancer". Gastroenterology 128 (5): 1160–71. doi:10.1053/j.gastro.2005.01.056.ببمد: 15887099.
- ^ ا ب to:a b Facista A, Nguyen H, Lewis C, Prasad AR, Ramsey L, Zaitlin B et al. (2012)."Deficient expression of DNA repair enzymes in early progression to sporadic colon cancer". Genome Integr 3 (1): 3. doi:10.1186/2041-9414-3-3. PMC 3351028.ببمد: 22494821.
- ^ ا ب ج د to:a b c d Chaisaingmongkol J, Popanda O, Warta R, Dyckhoff G, Herpel E, Geiselhart L et al. (2012). "Epigenetic screen of human DNA repair genes identifies aberrant promoter methylation of NEIL1 in head and neck squamous cell carcinoma". Oncogene 31 (49): 5108–16. doi:10.1038/onc.2011.660. ببمد: 22286769.
- ^ Fan CY (Mar 2004). "Epigenetic alterations in head and neck cancer: prevalence, clinical significance, and implications.". Curr Oncol Rep 6 (2): 152–61. doi:10.1007/s11912-004-0027-0. ببمد: 14751093.
- ^ Koutsimpelas D, Pongsapich W, Heinrich U, Mann S, Mann WJ, Brieger J (2012). "Promoter methylation of MGMT, MLH1 and RASSF1A tumor suppressor genes in head and neck squamous cell carcinoma: pharmacological genome demethylation reduces proliferation of head and neck squamous carcinoma cells". Oncol Rep 27 (4): 1135–41.doi:10.3892/or.2012.1624. ببمد: 22246327.
- ^ Sun W, Zaboli D, Liu Y, Arnaoutakis D, Khan T, Wang H et al. (2012). "Comparison of promoter hypermethylation pattern in salivary rinses collected with and without an exfoliating brush from patients with HNSCC". PLOS ONE 7 (3): e33642.doi:10.1371/journal.pone.0033642. ببمد: 22438973.
- ^ Puri SK, Si L, Fan CY, Hanna E. "Aberrant promoter hypermethylation of multiple genes in head and neck squamous cell carcinoma.". Am J Otolaryngol 26 (1): 12–7.doi:10.1016/j.amjoto.2004.06.007. ببمد: 15635575.
- ^ Zuo C, Zhang H, Spencer HJ, Vural E, Suen JY, Schichman SA et al. (2009). "Increased microsatellite instability and epigenetic inactivation of the hMLH1 gene in head and neck squamous cell carcinoma". Otolaryngol Head Neck Surg 141 (4): 484–490.doi:10.1016/j.otohns.2009.07.007. ببمد: 19786217.
- ^ Tawfik HM, El-Maqsoud NM, Hak BH, El-Sherbiny YM (2011). "Head and neck squamous cell carcinoma: mismatch repair immunohistochemistry and promoter hypermethylation of hMLH1 gene". Am J Otolaryngol 32 (6): 528–536. doi:10.1016/j.amjoto.2010.11.005.ببمد: 21353335.
- ^ Ai L, Vo QN, Zuo C, Li L, Ling W, Suen JY et al. (Jan 2004). "Ataxia-telangiectasia-mutated (ATM) gene in head and neck squamous cell carcinoma: promoter hypermethylation with clinical correlation in 100 cases.". Cancer Epidemiol Biomarkers Prev13 (1): 150–6. doi:10.1158/1055-9965.epi-082-3. ببمد: 14744748.
- ^ Nowak MA, Komarova NL, Sengupta A, Jallepalli PV, Shih I, Vogelstein B et al. (2002)."The role of chromosomal instability in tumor initiation". Proc Natl Acad Sci U S A 99(25): 16226–16231. doi:10.1073/pnas.202617399. PMC 138593. ببمد: 12446840.
- ^ ا ب Ballestar E (2010). "Epigenetics lessons from twins: prospects for autoimmune disease".Clin Rev Allergy Immunol 39 (1): 30–41. doi:10.1007/s12016-009-8168-4.ببمد: 19653134.
- ^ Wang LG, Chiao JW (September 2010). "Prostate cancer chemopreventive activity of phenethyl isothiocyanate through epigenetic regulation (review)". Int. J. Oncol. 37 (3): 533–9. doi:10.3892/ijo_00000702. ببمد: 20664922.
- ^ ا ب to:a b Iglesias-Linares A, Yañez-Vico RM, González-Moles MA (May 2010). "Potential role of HDAC inhibitors in cancer therapy: insights into oral squamous cell carcinoma". Oral Oncol.46 (5): 323–9. doi:10.1016/j.oraloncology.2010.01.009. ببمد: 20207580.
- ^ Spannhoff A, Sippl W, Jung M (January 2009). "Cancer treatment of the future: inhibitors of histone methyltransferases". Int. J. Biochem. Cell Biol. 41 (1): 4–11.doi:10.1016/j.biocel.2008.07.024. ببمد: 18773966.
- ^ Alfredo F Galvez, Na Chen, Janet Macasieb, and Ben O. de Lumen (October 15, 2001)."Chemopreventive Property of a Soybean Peptide (Lunasin) That Binds to Deacetylated Histones and Inhibits Acetylation". Cancer Research 61.
- ^ Dowden J, Hong W, Parry RV, Pike RA, Ward SG (April 2010). "Toward the development of potent and selective bisubstrate inhibitors of protein arginine methyltransferases".Bioorg. Med. Chem. Lett. 20 (7): 2103–5. doi:10.1016/j.bmcl.2010.02.069.ببمد: 20219369.
- ^ Kaminsky ZA, Tang T, Wang SC, Ptak C, Oh GH, Wong AH et al. (February 2009). "DNA methylation profiles in monozygotic and dizygotic twins". Nat. Genet. 41 (2): 240–5.doi:10.1038/ng.286. ببمد: 19151718.
- ^ O'Connor, Anahad (11 March 2008). "The Claim: Identical Twins Have Identical DNA". New York Times. Retrieved 2 May 2010.
- ^ Kaminsky ZA, Tang T, Wang SC, Ptak C, Oh GH, Wong AH et al. (2009). "DNA methylation profiles in monozygotic and dizygotic twins". Nat Genet 41 (2): 240–245.doi:10.1038/ng.286. ببمد: 19151718.
- ^ Casadesús J, Low D (September 2006). "Epigenetic gene regulation in the bacterial world". Microbiol. Mol. Biol. Rev. 70 (3): 830–56. doi:10.1128/MMBR.00016-06.PMC 1594586. ببمد: 16959970.
- ^ to:a b Jorg Tost (2008). Epigenetics. Norfolk, England: Caister Academic Press. ISBN 1-904455-23-9.
- ^ Davis BM, Chao MC, Waldor MK (2013). "Entering the era of bacterial epigenomics with single molecule real time DNA sequencing". Current Opinion in Microbiology 16 (2): 192–8. doi:10.1016/j.mib.2013.01.011. PMC 3646917. ببمد: 23434113.
- ^ Lluch-Senar M, Luong K, Lloréns-Rico V, Delgado J, Fang G, Spittle K et al. (2013). Richardson PM, ed. "Comprehensive Methylome Characterization of Mycoplasma genitalium and Mycoplasma pneumoniae at Single-Base Resolution". PLoS Genetics 9(1): e1003191. doi:10.1371/journal.pgen.1003191. PMC 3536716. ببمد: 23300489.
- ^ Murray IA, Clark TA, Morgan RD, Boitano M, Anton BP, Luong K et al. (2012). "The methylomes of six bacteria". Nucleic Acids Research 40 (22): 11450–62.doi:10.1093/nar/gks891. PMC 3526280. ببمد: 23034806.
- ^ Fang G, Munera D, Friedman DI, Mandlik A, Chao MC, Banerjee O et al. (2012). "Genome-wide mapping of methylated adenine residues in pathogenic Escherichia coli using single-molecule real-time sequencing". Nature Biotechnology 30 (12): 1232–9.doi:10.1038/nbt.2432. ببمد: 23138224.
وصلات خارجية
[عدل]- The Human Epigenome Project (HEP)
- The Epigenome Network of Excellence (NoE)
- Canadian Epigenetics, Environment and Health Research Consortium (CEEHRC)
- The Epigenome Network of Excellence (NoE)- public international site
- DNA Is Not Destiny – Discover Magazine cover story
- BBC – Horizon – 2005 – The Ghost In Your Genes
- Epigenetics article at Hopkins Medicine
- Towards a global map of epigenetic variation

